INDICE:
1. Introducción
2. Herramientas de estudio génico
3. Diabetes: genotipos y fenotipos
4. Complejidad genética de la DM2: QTL DM2 en humanos
5. Genes de riesgo para diabetes
2. Herramientas de estudio génico
3. Diabetes: genotipos y fenotipos
4. Complejidad genética de la DM2: QTL DM2 en humanos
5. Genes de riesgo para diabetes
6. TCF7L2 y DM2: Una relación especial
8. Epigenética
- Descubrimiento y estudios poblacionales
- Mecanismos propuestos para la influencia de TCF7L2
- La variante rs7903146
- Implicaciones clínicas
8. Epigenética
- Metilación de ADN
- Modificación de histonas
- ARN no codificante (ncARN)
9. Aspectos epigenéticos en la DM2: factores ambientales
- Alteraciones en los patrones de metilación del ADN
- Metilación diferencial del ADN en islotes de Langerhan en DM2
- Genes hipometilados/hipermetilados y activados en DM2
- Histonas ¿Memoria de la célula?
- Modificaciones en histonas en DM. Respuesta inflamatoria
- Modificación de histonas y desarrollo de células β
- Diabetes y ncARN
10. Dieta y epigenética
11. Influencia epigenética de la vida intrauterina
12. Herencia epigenética transgeneracional en DM2
REFERENCIAS
11. Influencia epigenética de la vida intrauterina
12. Herencia epigenética transgeneracional en DM2
REFERENCIAS
"Todo cambia; nada es" (Heráclito)
"Nada de lo que fue vuelve a ser, y las cosas y los hombres y los niños no son lo que fueron un día" (Ernesto Sábato)1. Introducción
En pacientes con Diabetes Mellitus Tipo 2 (DM2), además de las alteraciones en el metabolismo de la glucosa, frecuentemente se asocian obesidad, hipertensión, dislipemia, resistencia a la insulina y/o hiperinsulinemia. Estas anormalidades pueden tener causas moleculares y genéticas superpuestas, que complican aún más el diagnóstico y las opciones de tratamiento. En el curso de la enfermedad, muchos, pero no todos los pacientes, desarrollan comorbilidades, incluidas retinopatía, nefropatía, neuropatías y enfermedad cardiovascular. Actualmente no es posible evaluar el potencial de estas manifestaciones de la enfermedad desde el momento del diagnóstico inicial, lo que puede conducir a un manejo subóptimo de la DM2, aparición de complicaciones adicionales y mayores costos del tratamiento.
Las herramientas de estudio génico, como los estudios de ligamiento o linkage, los estudios de asociacion (GWAS o Genome-wide association study), el análisis de quantitative trait locus (QTL o eQTL) (en castellano expresión de cuantificación de rasgos por loci, que es una variación de los estudios de asociación) (*), los estudios de genes candidatos y los modelos animales, son valiosos en la identificación de nuevas vías involucradas en la regulación de la homeostasis de la glucosa. Así ha aumentado nuestra comprensión de las interacciones gen-gen y gen-ambiente, que impactan en la etiología y patogénesis de la DM2. Además, la identificación de variantes genéticas, que determinan las diferencias en la respuesta a los medicamentos antidiabéticos, será útil para evaluar una terapia farmacológica de primera línea para pacientes diabéticos.
La diabetes tipo 1 (DM1) es una enfermedad autoinmune en la que, principalmente, variaciones en genes imuno-reguladores predisponen a la destrucción inmuno-mediada de las células β por células T, en los islotes de Langerhans, en un proceso inflamatorio crónico (insulitis). En otro sentido, los estudios GWAS sugieren que la DM2 es, predominantemente, una enfermedad de la célula β, en la que, las alteraciones en genes que afectan a la función y/o masa de la célula β afectan a los mecanismos de compensación de las mismas, ante el aumento de la demanda de insulina. En ambas enfermedades se dan fuertes interacciones gen-ambiente, que desencadenan el proceso patogénico (en DM1 factores etiológicos virales y nutricionales no identificadas y en la DM2, principalmente, factores relacionados con el estilo de vida).
En la DM1, la inflamación de los islotes y la destrucción de células β, por mecanismos autoinmunes, son reconocidos desde hace tiempo como causas. Aunque sigue en discusión si los mecanismos moleculares, implicados en la lesión de la célula β, se relacionan predominantemente con inmunidad celular (células T citotóxicas) o con la acción de citoquinas, o con ambos mecanismos. En el caso de la DM2, han sido propuestos varios mecanismos: glucolipotoxicidad, alteración de la membrana celular por acúmulo de polipéptido amiloide en los islotes e inflamación (3).
3. Diabetes: genotipos y fenotipos
Se han identificado varios genes subyacentes en las formas monogénicas de diabetes y hay pruebas sólidas sobre el componente genético para la susceptibilidad a la DM2.
La diabetes monogénica, representa no mas del 5% de las diabetes no autoinmunes, e incluye formas en las que predomina la deficiencia de insulina y otras en las que predomina la resistencia grave a la misma. Se consideran diabetes monogénicas la diabetes tipo MODY, la diabetes neonatal (transitoria o permanente), la diabetes mitocondrial, las formas de diabetes asociadas a defectos del receptor de la insulina y las lipodistrofias familiares. En ellas es posible realizar un diagnóstico genético, lo que permite adelantar la respuesta al tratamiento y el pronóstico. La forma más frecuente de diabetes monogénica es la diabetes tipo MODY, que comprende siete subtipos que se caracterizan por un defecto en la secreción de insulina. Las formas de diabetes neonatal son causadas por mutaciones en los genes Kir6.2 y SUR. El diagnóstico genético permite individualizar el tipo de tratamiento de estas formas de diabetes (Tabla 1) (4,5).
Sin embargo, la forma poligénica de la diabetes es el resultado de la contribución de muchos genes, que interactúan con diferentes factores ambientales para producir una amplia variación en el curso clínico de la enfermedad. Los métodos experimentales, que han sido utilizados con éxito en la identificación de las bases genéticas de enfermedades monogénicas, como la enfermedad de Huntington o la fibrosis quística o las formas monogénicas de diabetes, no pueden ser aplicadas directamente para identificar la base génica de los rasgos complejos de la DM2. Todavía no se conoce cómo los genes interactúan entre sí y con el entorno para producir DM2. A diferencia de la DM1, donde el riesgo genético se concentra principalmente en la región HLA, el componente genético del riesgo de DM2 no se concentra en una sola región y parece ser el resultado de la interacción de múltiples genes diseminados por todo el genoma. Es posible que el componente genético de la DM2 se deba a múltiples variantes genéticas comunes de pequeño efecto (hipótesis de variante común de enfermedad común) (*), pero esto no es seguro y puede resultar que el efecto se deba a múltiples variantes raras o incluso a pocas variantes raras de gran efecto.
Frecuentemente las formas monogénicas de la diabetes son erróneamente diagnosticadas como DM2, cuando son entidades clínicas diferentes. En cualquier caso, se debe tener en cuenta que los límites entre las formas poligénicas y monogénicas no siempre se definen claramente a nivel genético. Los polimorfismos en genes implicados en formas monogénicas de diabetes también pueden jugar un papel en la DM2 o poligénica (7).
Basándose en resultados de una variedad de estudios poblacionales, familiares y de gemelos, se estima que la influencia de la herencia en la DM2 oscilan entre el 20 y el 80%. El riesgo de por vida de desarrollar DM2 es del 40% para las personas que tienenun padre con DM2 y del 70% si ambos padres se ven afectados. Los familiares de primer grado de individuos con DM2 tienen aproximadamente 3 veces más probabilidades de desarrollar la enfermedad que los individuos sin antecedentes familiares positivos de la enfermedad. La tasa de concordancia en los gemelos monocigóticos es de aproximadamente del 70%, mientras que la concordancia en los gemelos dicigóticos es solo del 20 al 30%. El riesgo familiar observado es mayor cuando los estudios se limitan a los padres en el rango de edad de 35-60 años, lo que indica el mayor papel desempeñado por los factores ambientales en aquellos que desarrollan diabetes en décadas mas tardías de la vida. Una proporción significativa de esta herencia refleja también los aspectos hereditarios de la obesidad, que es un importante factor impulsor de la DM2 en cualquier población.
5. Genes de riesgo para diabetes
Figura 4: Representación simplificada de las principales vías de transducción de señales en mamíferos.
Encuadrada en rojo señalamos la vía Wnt. By cybertory - This file was derived from: Signal transduction
v1.png, CC BY-SA 3.0, https://commons.wikimedia.org/w/index.php?curid=12081090
Figura 5: El panel superior resume los resultados del metanálisis incondicional. Los loci previamente establecidos se marcan en rojo y los loci identificados por el estudio actual se señalan en verde. Los genes utilizados para nombrar señales se han elegido en base a la proximidad al índice SNP. El panel inferior resume los resultados de un metaanálisis equivalente después del acondicionamiento en 30 SNP asociados a DM2 autosómicos previamente establecidos y recientemente identificados (indicados por las líneas punteadas debajo de estos loci en el panel superior). El nivel de significación de estos se indica con un punto naranja para niveles sugerentes (P<10-5) o de color púrpura en el caso de señales secundarias cercanas a loci ya relacionados con DM2 (P<10-4). En el panel superior destaca la evidencia rede asociación previamente establecida de TCF7L2. Voight BF, et al.. Twelve type 2 diabetes susceptibility loci identified through large-scale association analysis. Nat Genet. 2010; 42(7):579-89 (9).
Muchos de estos genes están activos en las células β o pueden estar involucrados en la secreción de insulina. Ello respalda la noción de que la disfunción de las células β es un paso final crucial en el camino hacia la diabetes. Sin embargo muy pocos de estos genes parecen desempeñar un papel en la sensibilidad a la insulina (aunque eso puede cambiar a medida que haya más información disponible) y los genes involucrados en la vía de señalización de la insulina rara vez aparecen en los estudios GWAS en DM2. Cuando se utilizaron los índices de la función de las células β (HOMA-B) y de sensibilidad a la insulina (HOMA-IR), derivadas de medidas de glucosa e insulina en ayunas de 37000 individuos, para tratar de identificar la función más afectada por varios genes de riesgo de DM2, se encontró que alelos de riesgo en diez loci (MTNR1B, SLC30A8, THADA, TCF7L2 ,KCNQ1, CAMK1D, CDKAL1, IGF2BP2, HNF1B y CENTD2) se asociaron (P<0.05) con una función reducida de células β, y solo tres loci (PPARG, FTO y KLF14) se asociaron con una sensibilidad a la insulina reducida. Además se observó que una alta proporción de loci de susceptibilidad DM2 alberga señales de asociación independientes que influyen en rasgos complejos aparentemente no relacionados. Una explicación para ello es que las variantes raras puedan tener un mayor efecto sobre la sensibilidad a la insulina o, tal vez, los factores ambientales juegan un papel más importante en la alteración de la sensibilidad a la insulina, que en la secreción de la misma (Figura 5).
Por otra parte, algunos de los genes que se encuentran asociados con DM2 también parecen estar relacionados con la dislipidemia, la enfermedad cardíaca aterosclerótica y el cáncer (8,9).
. TCF7L2 y DM2: Una relación especial
TCF7L2 se descubrió como un gen de susceptibilidad a la diabetes tipo 2 después de mapear una fuerte señal de enlace con el cromosoma 10q en una población mexicoamericana. Posteriormente, esta región se mapeó en la población islandesa y se confirmó en cohortes danesas y estadounidenses, donde se encontró que el locus de riesgo estaba ubicado en el intrón 3 del gen TCF7L2. Hay indicios de que este gen puede desempeñar un papel en el cáncer y en la diabetes. Otros estudios han demostrado que variantes genéticas no codificantes de TCF7L2 están asociadas con el riesgo de diabetes tipo 2 en poblaciones de ancestros diversos en el Reino Unido, los Países Bajos, Finlandia, Suecia, Francia, India y Japón.
Confirmado en el primer estudio GWAS de gran dimensión realizado en población francesa, después se sucedieron otros importantes estudios GWAS, incluido el estudio Welcome Trust, que genotipó 2000 sujetos con DM2, frente a 3000 controles. En este estudio se constató que TCF7L2 era la señal mas fuerte para DM2 con una OR de 1.36 en heterozigotos portadores del alelo de riesgo. Posteriormente los resultados de este estudio fueron reproducidos en la mayoría de las poblaciones estudiadas. Los portadores de los diversos alelos de riesgo identificados tienen un OR de 1.4 y los homocigotos pueden tener un OR de 2.5 (10, 11, 12).
Figura 6: Efectos metabólicos de diferentes genotipos en rs7903146 de TCF7L2 . ( A ) Correlación entre el área bajo la curva de insulina durante OGTT y IVGTT (cohorte de Botnia; n = 403). ( B ) Efecto incretina medido como 100% × (AUC ins OGTT - AUC ins IVGTT )/AUC ins OGTT en sujetos hiperglucémicos (glucosa plasmática en ayunas> 5.4 mmol / l) (cohorte Botnia; n = 235). ( C ) Correlación entre las concentraciones de GIP en ayunas (pg / ml) y glucagón (pg / ml) (cohorte de Botnia; n = 78). ( D ) Absorción de glucosa en todo el cuerpo durante una pinza euglucémica-hiperinsulinémica. ( E ) EGP basal. ( F) Cambio en el IMC con el tiempo en sujetos que se convirtieron a T2D. Las barras representan la media ± SEM. Las líneas azules representan portadores de genotipo sin riesgo y las líneas rojas de rs7903146 en TCF7L2. Lyssenko V. et al. J Clin Invest. 2007; 117(8):2155-63
Se ha confirmado una asociación muy significativa entre la susceptibilidad relacionada con la DM2 y las variantes comunes en el TCF7L2 en sujetos islandeses, y el resultado fue el mismo con el método de caso-control en cohortes danesas y estadounidenses. El alelo T del SNP rs7903146 aumentó el riesgo de DMT2 en la población francesa con 2367 casos y 2499 controles. Los mismos resultados fueron publicados en el caso de población japonesa, afroamericana y brasileña. Sin embargo, no se encontró asociación entre rs7903146 y DM2 en población china.
En cualquier caso, la calidad de los datos varía mucho en diferentes ensayos, lo que se considera una de las razones de los resultados inconsistentes, además de los pequeños tamaños de muestra. Para mejorar los resultados estadísticos se ha utilizado la estrategia de metaanálisis combinando muestras. En 2018 Weiyue Ding et al. realizaron un metanálisis de estudios publicados que incluían rs7903146 en relación con DM2 con el objetivo de lograr un resultado más completo. Se utilizaron un total de 28 estudios para reevaluar la asociación entre rs7903146 y DM2.
En total se incluyeron 56628 participantes (34232 casos y 22396 controles) de 28 estudios recogidos en 26 artículos de la literatura. El resultado de cuatro subgrupos (caucásico, asiático oriental, asiático meridional y otros) sugirió que rs7903146 se asocia significativamente con DM2 en todos los subgrupos y grupos valorados (15, 16, 17, 18, 19).
También se explora la posibilidad de que las variantes TCF7L2 pudieran servir como marcadores para adaptar regímenes terapéuticos personalizados. Por ejemplo, DPP4 es una peptidasa que degrada las incretinas como GLP-1. Los inhibidores DPP4 mejoran la secreción de insulina estimulada por incretina e inhiben la liberación de glucagón, lo que ayuda a normalizar los niveles de glucosa en sangre. Si se confirma una influencia negativa en la secreción de incretina como un eje patológico principal en la diabetes tipo 2 impulsada por expresion de variantes TCF7L2, los inhibidores de DPP4 podrían servir como contrapeso y revertir los efectos de las variantes de TCF7L2 en los niveles de incretina. Los polimorfismos TCF7L2 se han asociado con variaciones en la respuesta al tratamiento con sulfonilurea, pero estos efectos son probablemente demasiado modestos para dirigir el tratamiento.
Sin embargo, hay que hacer algunas consideraciones sobre la utilidad clínica de las variantes TCF7L2. En primer lugar, las variantes específicas de riesgo de DM2 de TCF7L2 varían de una población a otra. Se han observado tamaños de efectos similares en varios grupos étnicos para las variantes TCF7L2 más estudiadas, pero la investigación debe ampliarse más allá de las poblaciones no blancas. En segundo lugar, aunque los tamaños del efecto de las variantes de TCF7L2 son relativamente grandes, una imagen mucho más precisa de la estratificación del riesgo y la personalización terapéutica se basaría en una colección más amplia de genes de predisposición en lugar de TCF7L2 solo.
En cualquier caso, TCF7L2 podría ser un biomarcador prometedor para la prevención de enfermedades para aquellos sujetos con una elevada predisposición genética a la DM2. Pero no existe un papel clínico para el cribado de rutina para TCF7L2, y la presencia de variantes de TCF7L2 que están asociadas con un mayor riesgo de DM2, sino que debería considerarse en combinación con otros predictores conocidos, al evaluar el riesgo individual de un paciente para DM2 (20, 21, 22).
8. Epigenética
La epigenética estudia los mecanismos de expresión de los genes sin modificación de la secuencia de ADN de los mismos. Es decir, se refiere a cambios heredables, que abarcan un conjunto de reacciones químicas y otros procesos, capaces de modificar la actividad del ADN y, por tanto, la función de los genes, sin alterar la secuencia de nucleótidos que lo componen. Son conocidos en el proceso epigenético los mecanismos de metilación de bases, la modificación postraduccional de histonas (por procesos de acetilación, metilación, deaminación, isomerización de prolinas y ubiquitinización) y el ARN no codificante o ARN de interferencia (iARN), a los que nos referimos a continuación.
9. Aspectos epigenéticos en la DM2: factores ambientales
Los factores ambientales son fuente de modificaciones importantes en los genes. La regulación epigenética es responsable de los mecanismos adaptativos de los seres vivos. La plasticidad del genoma le concede la capacidad para ser modificado y modulado y el resultado es la formación de diferentes fenotipos, que responden a los diferentes factores ambientales. Estos cambios epigenéticos tienen un elevado grado de estabilidad y pueden ser heredables, manteniéndose activos en una línea celular durante varias generaciones. Pueden generarse fenotipos beneficiosos para la supervivencia, pero también se pueden producir errores en las modificaciones inducidas por el ambiente, que pueden heredarse y perdurar. Estos “errores” constituyen, en muchos casos condiciones para la aparición de fenotipos patológicos (34).
Dentro de la estructura molecular cromosómica existen tres niveles o capas: genes codificadores de proteínas, genes no codificadores y capa epigenética. El nivel epigenético resulta crucial para el desarrollo. En él se encuentran las claves del crecimiento, del envejecimiento y del desarrollo del cáncer y de otras enfermedades muy prevalentes. Los procesos epigéneticos, como ya hemos señalado, no cambian la secuencia de nucleótidos del ADN, pero influyen en la expresión del mismo.
El riesgo de desarrollar DM2 está muy influenciado por factores ambientales. Dado que nuestro código genético no cambia significativamente en una o dos generaciones, el incremento constante de prevalencia de la diabetes se debe principalmente a los cambios en el medio ambiente. Las epidemias de obesidad y DM2 son paralelas entre sí y la creciente prevalencia de ambas está relacionada con los cambios en los hábitos alimenticios y en el estilo de vida.
Los genes influyen de múltiples maneras en el riesgo de desarrollar DM2, pero no solo al alterar la secreción de insulina o la acción de la misma. También influyen sobre la forma en que cualquier individuo interactúa con los factores ambientales. La adopción de estilos de vida poco saludables y la disposición a cambiar dichos estilos de vida varía entre individuos de una misma población. La genética también influye en factores como el gusto y las preferencias alimentarias; así como sobre el mayor o menor gasto energético en reposo o el tipo de microbioma de cada individuo y, lo que es muy importante en la DM2, sobre su relación con el estilo de vida. Estas interacciones genético-ambientales pueden ser extremadamente complejas y son una de las razones por las que solo una pequeña proporción de la herencia de DM2 se ha podido explicar hasta la fecha.
Los avances en genética humana ha llevado a la identificación de un número relativamente grande de loci asociados a obesidad y DM2. Sin embargo, su contribución al riesgo de enfermedad es pequeña y su valor predictivo bajo, lo que sugiere que el estilo de vida juega un papel crucial en la obesidad y el desarrollo de DM2. Los estudios que investigan las interacciones entre el gen y el estilo de vida en la obesidad y la DM2 han sugerido que el efecto biológico de la predisposición genética puede ser parcial o totalmente abolido por la modificación del estilo de vida o el estilo de vida saludable y viceversa.
La importancia de los factores ambientales y de estilo de vida se ha puesto de manifiesto mediante estudios epidemiológicos, que han mostrado cómo el efecto negativo de varios genes, asociados con la obesidad y la DM2, puede atenuarse en individuos con niveles más altos de actividad física o estilos de vida saludables. Por el contrario, un patrón bajo de actividad física y dieta occidental (no en el caso de la dieta mediterránea) lo acentúa. Además, se ha descubierto que la actividad física, así como las intervenciones dietéticas y de estilo de vida combinadas, inducen una significativa disminución del peso corporal y de otros rasgos antropométricos, contribuyendo a abolir el riesgo de obesidad en portadores de alelos de riesgo de dos de los genes más fuertemente asociados con la obesidad: FTO y MC4R.
Con respecto a los resultados de DM2 en estudios a gran escala, hay pruebas sólidas de la mejora de los rasgos metabólicos y la atenuación del riesgo de diabetes entre los portadores de alelos de riesgo TCF7L2 mediante dieta y ejercicio. El alelo menor del gen PPARγ también se ha asociado con un riesgo sustancialmente mayor de DM2 y aterosclerosis. Sin embargo, se ha sugerido que la modificación del estilo de vida atenúa su efecto negativo sobre el perfil metabólico, el peso corporal y el riesgo de diabetes (36, 37).
Estudios experimentales han demostrado la influencia del ambiente intrauterino, por mecanismos epigenéticos, en el desarrollo de disfunción de células β. Es el caso de retraso del crecimiento intrauterino (intrauterin growth retardation o IUGR), en ratones, se pone en marcha un mecanismo epigenético mediado por una alteración en la acetilacion de histonas de nucleosomas relacionados con el gen Pdx1(*), que lleva a la represión de la expresión de dicho gen (Figura 19).
Uno de los objetivos actuales del tratamiento de la DM2 es la individualización. Se trata de superar el hecho de que los individuos con sintomatología diabética reciban similares tratamientos. Precisamente la capacidad de respuesta a los medicamentos, por sí sola, demuestra la heterogeneidad molecular y de estilo de vida en la DM2. La optimización de los medicamentos para cada paciente puede ser, en la actualidad, un proceso largo de prueba y error, que implica una cantidad significativa de tiempo y un gasto considerable.
La complejidad etiológica, subyacente a la DM2, ha dificultado la identificación de los genes implicados en el desarrollo de la enfermedad. Debido a la variabilidad en el curso clínico y en la capacidad de respuesta a las terapias farmacológicas, la identificación y caracterización de las variantes genéticas subyacentes será importante en el futuro en el desarrollo de un tratamiento individualizado.
2. Herramientas de estudio génico
 |
Figura
2:
Elementos de la cromatina: Cromosomas,
cromátides, telómeros, centrómeros, nucleosomas, histonas, cadenas de ADN, bases (adenina, citosina, guanina, timidina) |
(*) Los estudios de ligamiento identifican regiones cromosómicas con alta probabilidad de albergar variantes implicadas en la aparición de un trastorno. El ligamiento es la tendencia de los genes y otros marcadores genéticos a heredarse juntos debido a su ubicación cerca uno del otro en el mismo cromosoma. A grandes rasgos, en un estudio de ligamiento se evalúan marcadores espaciados a lo largo del genoma en los miembros de una o varias familias con más de un individuo afecto y se investiga si alguno de los alelos de los marcadores cosegrega con la enfermedad a través de las genealogías. Esta estrategia es eficaz si el riesgo de la variante o variantes causales es elevado y si existen genes de efecto mayor en los que se localizan las variantes de susceptibilidad. Los estudios de ligamiento pierden sensibilidad si existe heterogeneidad de locus (existencia de varios loci implicados en la enfermedad), frente a la heterogeneidad alélica (existencia en el mismo gen de distintos alelos que causan susceptibilidad a la patología).
Un estudio de asociación del genoma completo o GWAS (Acrónimo del ingles: Genome-wide association study) o WGAS (Whole genome association study) es un análisis de una variación genética, a lo largo de todo el genoma humano, para identificar su asociación a un rasgo observable. Los GWAS suelen centrarse en asociaciones entre los polimorfismos de un solo nucleótido (SPN) y rasgos como las principales enfermedades. Para ello se deben estudiar una gran cantidad de individuos, de modo que se puedan comparar datos genéticos entre ellos. De este modo, al comparar los datos genéticos obtenidos de las secuenciaciones de los genomas de diferentes individuos, podemos hallar genes presuntamente ligados a enfermedades o caracteres. Por ejemplo, podemos contrastar cómo quizás se produce la aparición de uno o varios SNPs (variación de un solo par de bases) o una deleción, repetición, etc., en una secuencia del genoma siempre que aparece el mismo fenotipo, pudiendo así concluir que este cambio a nivel genético es probable que se corresponda con un rasgo. En líneas generales, en un estudio de asociación caso-control se pretende la identificación de variantes alélicas que están presentes en individuos con un fenotipo concreto a una frecuencia significativamente diferente que en individuos sin ese fenotipo.
El genotipado de SNP es la medición de variaciones genéticas de polimorfismos de un solo nucleótido entre miembros de una especie. Es de los tipos más comunes de variación genética y consiste en la mutación de un solo par de bases en un locus específico, que generalmente consta de dos alelos. Los SNP están involucrados en la etiología de muchas enfermedades humanas y han adquirido un interés particular en la farmacogenética. Debido a que los SNP se conservan durante la evolución, se han propuesto como marcadores para su uso en análisis de loci de rasgos cuantitativos (QTL) y en estudios de asociación en lugar de microsatélites.
GWAS y eQTLs. Existen millones de variantes genéticas y la interpretación de sus efectos funcionales en los caracteres humanos es uno de los mayores retos en la genómica. Los eQTLS son loci en el DNA que actúan como elementos regulatorios de la expresión génica, la variación de estos eQTLs y sus consecuencias a nivel de expresión añade una dimensión funcional a nuestros conocimientos, la cual puede ser usada en estudios de enfermedades. Una proporción de las variantes descritas en los GWAS son eQTLs o trQTLs, por lo que, la integración del genoma secuenciado, eQTLs y el fenotipo celular ayuda a comprender los genes causales de la enfermedad, las variantes genéticas causales que subyacen a los GWAS y los procesos biológicos que intervienen en estos.
Los estudios de ligamiento buscan identificar loci que cosegregan con el rasgo a través de la genealogia familiar, mientras que los estudios de asociación buscan identificar variantes particulares que están asociadas con el fenotipo a nivel de población. Son métodos complementarios que, juntos, proporcionan medios para sondear el genoma y describir la etiología de rasgos humanos complejos. En estudios de ligamiento, buscamos identificar los loci que cosegregan con una región genómica específica, marcada por marcadores polimórficos, dentro de las familias. En los estudios de asociación, buscamos una correlación entre una variación genética específica y la variación del rasgo en la muestra de individuos, lo que implica un papel causal de la variante (1,2).
 |
Tabla
1: Tipos de diabetes monogénicas. (R.
Barrio.
Diabetes
monogénicas: enfoque diagnóstico y tipos
más frecuentes. Av
Diabetol.
2007;
23(5):333-340).
|
En la DM1, la inflamación de los islotes y la destrucción de células β, por mecanismos autoinmunes, son reconocidos desde hace tiempo como causas. Aunque sigue en discusión si los mecanismos moleculares, implicados en la lesión de la célula β, se relacionan predominantemente con inmunidad celular (células T citotóxicas) o con la acción de citoquinas, o con ambos mecanismos. En el caso de la DM2, han sido propuestos varios mecanismos: glucolipotoxicidad, alteración de la membrana celular por acúmulo de polipéptido amiloide en los islotes e inflamación (3).
Se han identificado varios genes subyacentes en las formas monogénicas de diabetes y hay pruebas sólidas sobre el componente genético para la susceptibilidad a la DM2.
La diabetes monogénica, representa no mas del 5% de las diabetes no autoinmunes, e incluye formas en las que predomina la deficiencia de insulina y otras en las que predomina la resistencia grave a la misma. Se consideran diabetes monogénicas la diabetes tipo MODY, la diabetes neonatal (transitoria o permanente), la diabetes mitocondrial, las formas de diabetes asociadas a defectos del receptor de la insulina y las lipodistrofias familiares. En ellas es posible realizar un diagnóstico genético, lo que permite adelantar la respuesta al tratamiento y el pronóstico. La forma más frecuente de diabetes monogénica es la diabetes tipo MODY, que comprende siete subtipos que se caracterizan por un defecto en la secreción de insulina. Las formas de diabetes neonatal son causadas por mutaciones en los genes Kir6.2 y SUR. El diagnóstico genético permite individualizar el tipo de tratamiento de estas formas de diabetes (Tabla 1) (4,5).
Sin embargo, la forma poligénica de la diabetes es el resultado de la contribución de muchos genes, que interactúan con diferentes factores ambientales para producir una amplia variación en el curso clínico de la enfermedad. Los métodos experimentales, que han sido utilizados con éxito en la identificación de las bases genéticas de enfermedades monogénicas, como la enfermedad de Huntington o la fibrosis quística o las formas monogénicas de diabetes, no pueden ser aplicadas directamente para identificar la base génica de los rasgos complejos de la DM2. Todavía no se conoce cómo los genes interactúan entre sí y con el entorno para producir DM2. A diferencia de la DM1, donde el riesgo genético se concentra principalmente en la región HLA, el componente genético del riesgo de DM2 no se concentra en una sola región y parece ser el resultado de la interacción de múltiples genes diseminados por todo el genoma. Es posible que el componente genético de la DM2 se deba a múltiples variantes genéticas comunes de pequeño efecto (hipótesis de variante común de enfermedad común) (*), pero esto no es seguro y puede resultar que el efecto se deba a múltiples variantes raras o incluso a pocas variantes raras de gran efecto.
(*) Hipótesis enfermedad común/variante común. El hecho de que las enfermedades complejas fuesen comunes en la población hizo pensar que la razón de esta alta prevalencia podría ser que los factores genéticos de predisposición estuviesen presentes con relativa frecuencia en las poblaciones humanas. En base a esta idea se formuló la hipótesis enfermedad común / variante común (Lander, 1996). Eric S. Lander. The New Genomics: Global Views of Biology. Science 1996; 274,(5287):536-539 (6).
 |
Figura
3:
En las formas poligénicas (DM2) los factores ambientales cobran una
importancia fundamental sobre la base genética
|
El agrupamiento del riesgo de DM2, encontrado en varios estudios familiares no se debe enteramente a factores genéticos. Los procesos epigenéticos pueden producir un riesgo hereditario durante una o varias generaciones. Factores intrauterinos y relacionados con el embarazo pueden afectar al riesgo en hermanos y el ambiente compartido puede ser difícil de controlar en muchos de estos estudios. Por lo tanto, el componente genético de la DM2 puede ser inferior al estimado en estudios anteriores. Mas adelante en este artículo abordamos los aspectos epigenéticos relacionados con la DM2.
Las formas clínicas poligénicas de DM2 se inician en edad tardía y se manifiestan tanto con alteraciones de la secreción de insulina, como de la sensibilidad tisular a la misma. Su cuadro clínico procede de la interacción de factores ambientales y genéticos, frecuentemente con polimorfismos de muchos genes. Estos polimorfismos puede estar localizados en la codificación o en la regulación o expresión de los genes y estar presentes, aunque con diferentes frecuencias, en pacientes con DM2, así como en poblaciones sanas. Diferencias en la secuencia en unos pocos genes se han asociado, hasta ahora, con formas complejas, poligénicas, de la DM2, que afectan por ejemplo, a la expresión de proteinas como calpaína 10, PPARγ (peroxisome proliferator-activated receptor-γ), KCJN11 (potassium channel, inwardly rectifying subfamily J, member 11) y la misma insulina. Además, existe alguna evidencia de que los genes relacionados con la adiponectina, el IRS-1 y algunos otros, también pueden influir en la susceptibilidad a desarrollar DM2. Pero son muchos más los genes candidatos o ya claramente relacionados con el polimorfismo de la enfermedad y que han sido estudiados como candidatos a tener un papel relevante en la herencia de la DM2 (Tabla 2).
El análisis de eQTL, puede identificar regiones de cromosomas que contribuyen a un rasgo complejo. Los QTL se encuentran mediante el análisis estadístico de la frecuencia con la que una región de un cromosoma se asocia con un fenotipo medible. En el caso de la DM2, en que se valoran rasgos como los niveles de insulina o la respuesta de la misma a los niveles de glucosa, un QTL puede contribuir con un 20% al rasgo, mientras que otro puede contribuir solo con un 1%. Las contribuciones variarán dependiendo de las interacciones entre genes y nutrientes para el gen responsable de la QTL y si ese gen interactúa con otros genes en el genoma. La suma de las contribuciones de alelos causales en diferentes QTL produce el rasgo o enfermedad específica. El concepto de múltiples genes y múltiples influencias ambientales contribuyendo a un rasgo complejo sería el paradigma de lo que se sabe actualmente sobre la genómica de la DM2.
El análisis de eQTL, puede identificar regiones de cromosomas que contribuyen a un rasgo complejo. Los QTL se encuentran mediante el análisis estadístico de la frecuencia con la que una región de un cromosoma se asocia con un fenotipo medible. En el caso de la DM2, en que se valoran rasgos como los niveles de insulina o la respuesta de la misma a los niveles de glucosa, un QTL puede contribuir con un 20% al rasgo, mientras que otro puede contribuir solo con un 1%. Las contribuciones variarán dependiendo de las interacciones entre genes y nutrientes para el gen responsable de la QTL y si ese gen interactúa con otros genes en el genoma. La suma de las contribuciones de alelos causales en diferentes QTL produce el rasgo o enfermedad específica. El concepto de múltiples genes y múltiples influencias ambientales contribuyendo a un rasgo complejo sería el paradigma de lo que se sabe actualmente sobre la genómica de la DM2.
4. Complejidad genética de la DM2: QTL DM2 en humanos
Mediante análisis de expresión de QTLs se han aislado siete QTL, con un LOD>3.6 (límite de detección, como medida de significancia), que contribuyen al desarrollo de DM2. Dichos loci se detectaron en grupos ancestrales y específicos en los cromosomas 1q25.3, 2q37.3, 3p24.1, 3q28, 10q26.13, 12q24.31 y 18p11.22. Ello implica una enorme complejidad potencial, a un nivel que podríamos imaginar si multiplicamos el número de alelos posibles por cada QTL. Sin embargo, en poblaciones humanas, el número real de combinaciones no es tan elevado como el que resultaría de dicho cálculo, por la variación de la frecuencia de alelos entre grupos ancestrales. Lo que hace probable, por ejemplo, que cromosomas de ascendencia europea tengan una proporción diferente de alelos en un locus determinado a los cromosomas de ascendencia africana. Sin embargo no se conocen dichos genes, ni su contribución cuantitativa en diferentes grupos ancestrales. Tampoco se conoce la combinación de alelos en los siete loci, que significan diferentes edades de inicio y de severidad de la DM2.
Para mayor complejidad, además de los siete loci citados con un LOD>3.6, se han identificado otros diecisiete QTL, en diversas regiones de los cromosomas 1, 2, 4, 5, 7, 8, 9, 10, 11, 12, 20 y X, con una significancia menor (LOD, entre 2.0 y 3.6), que podrían considerarse “sospechosos” de contribuir a la DM2 (8).
Mediante análisis de expresión de QTLs se han aislado siete QTL, con un LOD>3.6 (límite de detección, como medida de significancia), que contribuyen al desarrollo de DM2. Dichos loci se detectaron en grupos ancestrales y específicos en los cromosomas 1q25.3, 2q37.3, 3p24.1, 3q28, 10q26.13, 12q24.31 y 18p11.22. Ello implica una enorme complejidad potencial, a un nivel que podríamos imaginar si multiplicamos el número de alelos posibles por cada QTL. Sin embargo, en poblaciones humanas, el número real de combinaciones no es tan elevado como el que resultaría de dicho cálculo, por la variación de la frecuencia de alelos entre grupos ancestrales. Lo que hace probable, por ejemplo, que cromosomas de ascendencia europea tengan una proporción diferente de alelos en un locus determinado a los cromosomas de ascendencia africana. Sin embargo no se conocen dichos genes, ni su contribución cuantitativa en diferentes grupos ancestrales. Tampoco se conoce la combinación de alelos en los siete loci, que significan diferentes edades de inicio y de severidad de la DM2.
Para mayor complejidad, además de los siete loci citados con un LOD>3.6, se han identificado otros diecisiete QTL, en diversas regiones de los cromosomas 1, 2, 4, 5, 7, 8, 9, 10, 11, 12, 20 y X, con una significancia menor (LOD, entre 2.0 y 3.6), que podrían considerarse “sospechosos” de contribuir a la DM2 (8).
5. Genes de riesgo para diabetes
Los estudios de ligamiento, solo han identificado dos genes claramente asociados al desarrollo de la DM2: calpaina 10 (CAPN10) y el factor de transcripción 7 similar a 2 (TCF7L2):
CAPN10 fue el primer gen DM2 descubierto por análisis de ligamiento cuando un locus en el cromosoma 2 se asoció con DM2. Después, grandes metaanálisis han demostrado la probable asociación de variantes de CAPN10 con DM2.
TCF7L2 demostró susceptibilidad a DM2 tras mapeo de una señal de enlace al cromosoma 10q, que fue confirmada en diversas poblaciones: mexicoamericana e islandesa, así como en cohortes danesas y norteamericanas, donde se encontró que el locus de riesgo estaba ubicado en el intrón 3 del gen TCF7L2. La asociación entre DM2 y varios polimorfismos de un solo nucleótido (SNP) en el gen TCF7L2 se ha confirmado desde entonces en múltiples estudios GWAS, en diferentes grupos étnicos. Este gen sigue siendo más fuertemente asociado al riesgo de padecer DM2.El estudio de genes candidatos se ha basado en profundizar en conocimiento de genes que ya se sabían relacionados con la DM2, mediante secuenciación focal. Los candidatos eran genes que se relacionaban con el metabolismo de la glucosa, con la secreción de insulina, con los receptores de la misma o con la señalización posterior al receptor. Sin embargo, muchos de los genes relacionados con estas funciones, no demostraron asociación con el desarrollo de DM2 en la población, aunque algunos de ellos tenían un mayor significado en la enfermedad o en alguna de sus variedades monogénicas:
PPARG resulta una diana interesante por la posible asociación de sus mutaciones con la DM2. Este gen codifica los receptores ppARγ, relacionados con la sensibilidad celular a la insulina, por otra parte, objetivo molecular de una clase de fármacos: las tiazolidendionas (roxiglitazona y pioglitazona). En este sentido, se observó que un cambio de prolina a arginina en la posición 12 del gen PPARG generaba un aumento del 20% en el riesgo de desarrollar DM2. Otros polimorfismos de este gen también tienen un papel en algunos casos de diabetes.
KCNJ11 codifica el canal de potasio sensible a ATP Kir6.2. Interviene en la regulación de la secreción de insulina por las células β . Las mutaciones en este gen son causa conocida de diabetes neonatal. Pero además un polimorfismo de KCNJ11 está asociado a DM2. El cociente de riesgo u odd ratio (OR) de desarrollar DM2 es de 1.2 en portadores del alelo de riesgo, que además está asociado a disminución de la secreción de insulina en algunas poblaciones.
Mutaciones raras de alta penetrancia de los genes HNF1A, HNF1B y HNF4A tienen una clara relación con la diabetes monogénica en sujetos jóvenes (diabetes tipo MODY). Algunas variantes de estos genes que no conducen a MODY se asocian a disminución de la secreción de insulina y aumento de riesgo de DM2 en algunas poblaciones. Aunque su repercusión en la población general es escasa.Los estudios de asociación (GWAS) han permitido, mediante la tecnología de genotipado SNP de alto rendimiento y la disponibilidad de datos de HapMap (*), escanear cientos de miles de SNP que estaban en desequilibrio de enlace con millones de SNP en todo el genoma. En los últimos años, el número de variantes conocidas para DM2 ha aumentado significativamente, incluyendo la confirmación de las variantes identificadas anteriormente por los genes candidatos y los estudios de ligamiento:
(*) HapMap fue un proyecto Internacional con el objetivo de desarrollar un mapa de haplotipos del genoma humano, para describir los patrones comunes de variación genética humana. HapMap se utiliza para encontrar variantes genéticas que afectan a la salud, a la enfermedad y las respuestas a las drogas y los factores ambientales. La información producida por el proyecto se pone a disposición gratuitamente para la investigación.
TCF7L2 ya había sido identificado mediante estudios de ligamiento y resultó ser la señal mas significativa y replicada detectada también en los estudios GWAS. Sigue siendo el gen con evidencia de riesgo de DM2 más robusta, de los identificados hasta la fecha. Los portadores de los diversos alelos de riesgo identificados tienen un OR de 1.4 y los homocigotos pueden llegar a un OR de 2.5. Este gen codifica un factor de transcripción que interviene en la vía de señalización Wnt (*) y está activo en células β. La significancia del mismo y la importante actividad investigadora desarrollada en relación con TCF7L2 hace que le dediquemos un apartado independiente mas adelante.
(*) Las vías de señalización de Wnt (acrónimo creado a partir de los nombres Wingless e Integration-1) son un grupo de sistemas de transducción de señales mediante proteínas, que pasan señales a una célula, a través de los receptores de la superficie celular. Las vías de señalización de Wnt utilizan la comunicación célula-célula cercana (paracrina) o la comunicación en la misma célula (autocrina). Estas vías están altamente conservadas evolutivamente en todos los animales. Existen tres rutas de señalización de Wnt: la ruta de Wnt canónica, la ruta de polaridad celular plana no canónica y la ruta de Wnt/calcio no canónica.
 |
Encuadrada en rojo señalamos la vía Wnt. By cybertory - This file was derived from: Signal transduction
v1.png, CC BY-SA 3.0, https://commons.wikimedia.org/w/index.php?curid=12081090
Las tres vías se activan por la unión de una proteína Wnt-ligando de la familia de receptores Frizzled (familia de proteínas receptoras acopladas a proteínas G, que sirven como receptores en la vía Wnt y de otras vías de señalización), desde donde pasa la señal biológica al citosol a la proteína Dishevelled (Dsh) (una fosfoproteína citoplasmática que actúa directamente mas allá de los receptores Frizzled). La vía Wnt canónica conduce a la regulación de la transcripción génica, y se cree que está regulada negativamente, en parte, por el gen SPATS1. La vía de polaridad de la célula plana no canónica regula el citoesqueleto responsable de la forma celular. La vía no canónica Wnt/calcio regula el calcio dentro de la célula (Fig. 4).
HHEX (homeobox expresado hematopoyéticamente) fue identificado como otro de los genes asociados a DM2 en estudios de poblaciones caucásicas y asiáticas. Ubicado también en el cromosoma 10q, codifica un factor de transcripción involucrado también en la señalización Wnt. Los alelos de riesgo parecen conferir un OR de desarrollar T2D de 1.5.
SLC30A8 (portador de solutos familia 30 (transportador de zinc), miembro 8) codifica una proteína involucrada en almacenamiento y secreción de gránulos de insulina, que se expresa a un alto nivel solo en el páncreas, particularmente en los islotes de Langerhans. Dicho mecanismo podría implicar que este gen estuviera involucrado en un mayor riesgo de diabetes tipo DM2. Dicha asociación que se ha testado en diferentes poblaciones y también se ha señalado asociado con DM1.
CDKN2 A/B (inhibidor de quinasa dependiente de ciclina 2 A/B). Estos genes se encuentran en el cromosoma 9p21 y generan ciertas variantes de transcripción. Ambos genes son importantes reguladores del ciclo celular con un papel en la supresión tumoral. Esta región se ha mostrado asociada con DM2, en múltiples estudios de GWAS, en diferentes poblaciones. Los alelos de riesgo de estos genes confieren un OR para el desarrollo de DM2 de entre 1.2 y 1.5. Todo parece indicar que las mutaciones de estos genes juegan un papel en la secreción de insulina.
IGF2BP2 (proteína 2 de unión al ARNm del factor de crecimiento similar a la insulina 2) codifica una proteína que se une a la 5'-UTR (del inglés untranslated region o región no traducida) (*) del ARNm del factor de crecimiento similar a la insulina 2 (IGF2) y, por lo tanto, regula la traducción del IGF2. Se han caracterizado variantes alternativas, que codifican diferentes isoformas. Además este gen está asociado con el riesgo de DM2 en múltiples estudios de asociación. Como las variantes de los genes HHEX y CDKN2A/B, puede tener un papel en la función de las células β.
(*) Se denomina región no traducida o UTR (del inglés untranslated region o bien untranslated trailer) al sector extremo de los genes. Se habla generalmente de una región no traducida cinco prima (5'-UTR en inglés) y de una región no traducida tres prima (3'-UTR en inglés) que son las dos partes no traducidas de cada gen, debido a que se encuentran colindando con el marco abierto de lectura (u ORF). Las UTR poseen gran importancia en la regulación de la expresión génica. Por ejemplo, existen proteínas adaptadoras que reconocen secuencias específicas, no codificantes, del 3'-UTR. Además, están implicadas en la correcta expresión espacial y temporal de los genes.Como puede verse en la Tabla 2, los genes que, de alguna manera, se relacionan con la herencia poligénica de la DM2 constituyen una nutrida lista y esta no ha parado de crecer en las últimas décadas (mas de un centenar en la actualidad). Y, sin embargo, como también puede apreciarse en dicho listado, los OR individuales de los alelos de riesgo son, por lo general, menores de 1.3. Las excepciones mas notables las constituyen los genes TCF7L2 y KCNQ1.
 |
Muchos de estos genes están activos en las células β o pueden estar involucrados en la secreción de insulina. Ello respalda la noción de que la disfunción de las células β es un paso final crucial en el camino hacia la diabetes. Sin embargo muy pocos de estos genes parecen desempeñar un papel en la sensibilidad a la insulina (aunque eso puede cambiar a medida que haya más información disponible) y los genes involucrados en la vía de señalización de la insulina rara vez aparecen en los estudios GWAS en DM2. Cuando se utilizaron los índices de la función de las células β (HOMA-B) y de sensibilidad a la insulina (HOMA-IR), derivadas de medidas de glucosa e insulina en ayunas de 37000 individuos, para tratar de identificar la función más afectada por varios genes de riesgo de DM2, se encontró que alelos de riesgo en diez loci (MTNR1B, SLC30A8, THADA, TCF7L2 ,KCNQ1, CAMK1D, CDKAL1, IGF2BP2, HNF1B y CENTD2) se asociaron (P<0.05) con una función reducida de células β, y solo tres loci (PPARG, FTO y KLF14) se asociaron con una sensibilidad a la insulina reducida. Además se observó que una alta proporción de loci de susceptibilidad DM2 alberga señales de asociación independientes que influyen en rasgos complejos aparentemente no relacionados. Una explicación para ello es que las variantes raras puedan tener un mayor efecto sobre la sensibilidad a la insulina o, tal vez, los factores ambientales juegan un papel más importante en la alteración de la sensibilidad a la insulina, que en la secreción de la misma (Figura 5).
Por otra parte, algunos de los genes que se encuentran asociados con DM2 también parecen estar relacionados con la dislipidemia, la enfermedad cardíaca aterosclerótica y el cáncer (8,9).
. TCF7L2 y DM2: Una relación especial
- Descubrimiento y estudios poblacionales
TCF7L2 se descubrió como un gen de susceptibilidad a la diabetes tipo 2 después de mapear una fuerte señal de enlace con el cromosoma 10q en una población mexicoamericana. Posteriormente, esta región se mapeó en la población islandesa y se confirmó en cohortes danesas y estadounidenses, donde se encontró que el locus de riesgo estaba ubicado en el intrón 3 del gen TCF7L2. Hay indicios de que este gen puede desempeñar un papel en el cáncer y en la diabetes. Otros estudios han demostrado que variantes genéticas no codificantes de TCF7L2 están asociadas con el riesgo de diabetes tipo 2 en poblaciones de ancestros diversos en el Reino Unido, los Países Bajos, Finlandia, Suecia, Francia, India y Japón.
Confirmado en el primer estudio GWAS de gran dimensión realizado en población francesa, después se sucedieron otros importantes estudios GWAS, incluido el estudio Welcome Trust, que genotipó 2000 sujetos con DM2, frente a 3000 controles. En este estudio se constató que TCF7L2 era la señal mas fuerte para DM2 con una OR de 1.36 en heterozigotos portadores del alelo de riesgo. Posteriormente los resultados de este estudio fueron reproducidos en la mayoría de las poblaciones estudiadas. Los portadores de los diversos alelos de riesgo identificados tienen un OR de 1.4 y los homocigotos pueden tener un OR de 2.5 (10, 11, 12).
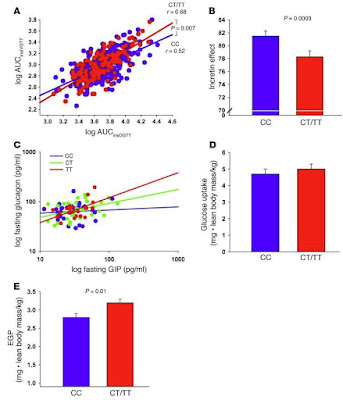 |
El estudio de los mecanismos por los que la expresión de TCF7L2 se asocia a un aumento del riesgo de DM2 y a una función alterada de las células beta, mostró que, el mayor riesgo de DM2, conferido por las variantes en TCF7L2, implica el eje entero-insular, la expresión mejorada del gen en los islotes y la secreción de insulina alterada, efectos incretínicos y mayor tasa de producción de glucosa hepática. A esa conclusión se llegó tras constatar en un estudio prospectivo la capacidad de las variantes comunes del gen de predecir una futura DM2. Para ello se estudiaron en escandinavos, seguidos durante 22 años y genotipados para tres SNP (rs7903146, rs12255372 y rs10885406) en TCF7L2 (2 grandes cohortes prospectivas de 9.663 individuos de Suecia y Finlandia). En un subconjunto de estos individuos se exploró la correlación genotipo-fenotipo, mediante el estudio de la influencia en los niveles de insulina, glucagón y GIP; así como sus efectos incretínicos y sobre la sensibilidad hepática y en tejidos periféricos a la insulina. Lamentablemente no se pudo cuantificar el efecto sobre incretinas tipo GLP1 aunque si en GIP. En la cohorte finlandesa se valoró, además, la relación genotipo-fenotipo en cuanto a IMC y niveles de leptina. Utilizando un vector viral sobre islotes in vitro se observó que la sobreexpresión en este tejido pancreático de TCF7L2 reducía la secreción de insulina estimulada por glucosa (13).
|
El TCF7L2 es un candidato a ser tenido muy en cuenta en la etiopatogenia de la DM2. Ello se sustenta en su expresión clara en tejido pancreático y hepático, así como por la relación de sus variantes con la DM2 en numerosos grupos étnicos de todo el mundo. Como hemos señalado antes, en el caso de las variantes asociadas, el riesgo de DM2 aumenta 1,5 veces en heterocigotos y hasta 2,5 veces en homocigotos. Ello representa un riesgo atribuible en la población del 21%, que hace que las variantes TCF7L2 sean los factores de riesgo genéticos más fuertes conocidos para la DM2.
Del gen TCF7L2 han sido identificados un cierto número de loci, como el rs10749127, rs10787475, rs11196224, rs12775879, rs17130188, rs290481, rs290487, rs290489, rs3750804, rs4918792, rs6585194, rs7085532, rs7094463, rs7919409, rs966227, rs7903146, rs11196205 y rs12255372 que representan su polimorfismo. Entre estos polimorfismos de un solo nucleótido (SNP), el TCF7L2 rs7903146 muestra la asociación más fuerte con la diabetes tipo 2.
La incidencia de diabetes tipo 2 en las mujeres afroamericanas es más del doble que la de las mujeres blancas en los Estados Unidos. Sin embargo, si bien se han descubierto más de 75 loci genéticos para la diabetes tipo 2 en poblaciones de ascendencia europea, asiática y mexicana, solo se han descubierto 3 variantes nuevas en estudios de asociación de todo el genoma (GWAS) de poblaciones de ascendencia africana; la más notable es SNP TCF7L2 rs7903146, la variante más significativamente asociada con la diabetes tipo 2 hasta la fecha.
TCF7L2 es un factor de transcripción y un componente clave de la vía de señalización Wnt (del que ya hemos hablado anteriormente), y está involucrado en el desarrollo de una amplia variedad de linajes y órganos celulares. Los mecanismos potenciales a través de los cuales las variantes de TCF7L2 influyen en la DM2 incluyen su papel en la adipogénesis, la miogénesis y el desarrollo de islotes pancreáticos, así como en la supervivencia de las células beta y la función del gránulo secretor de insulina. La proteína TCF7L2 aumenta durante la adipogénesis en las células 3T3-L1 y las células madre primarias de adipocitos. La inactivación de la proteína TCF7L2 mediante la eliminación del dominio de unión al ADN del grupo de alta movilidad (HMG), en los adipocitos maduros in vivo, conduce a intolerancia a la glucosa en todo el cuerpo y resistencia a la insulina hepática. Este fenotipo está asociado con una mayor masa de tejido adiposo subcutáneo, hipertrofia de adipocitos e inflamación. También participa en la regulación transcripcional de los genes para el proglucagón y de los péptidos similares al glucagón GLP-1 y GLP-2, que juegan un papel transcendente en la secreción de insulina posprandial.
Los polimorfismos TCF7L2 se han asociado con una secreción alterada de insulina, a través de efectos directos sobre las células beta de los islotes pancreáticos. De hecho, la desregulación del metabolismo de la glucosa, la disminución del procesamiento de la proinsulina y los niveles elevados de GIP y HbA1c se observan incluso en individuos normoglucémicos, con polimorfismos TCF7L2, antes del inicio de la DM2.
Del gen TCF7L2 han sido identificados un cierto número de loci, como el rs10749127, rs10787475, rs11196224, rs12775879, rs17130188, rs290481, rs290487, rs290489, rs3750804, rs4918792, rs6585194, rs7085532, rs7094463, rs7919409, rs966227, rs7903146, rs11196205 y rs12255372 que representan su polimorfismo. Entre estos polimorfismos de un solo nucleótido (SNP), el TCF7L2 rs7903146 muestra la asociación más fuerte con la diabetes tipo 2.
La incidencia de diabetes tipo 2 en las mujeres afroamericanas es más del doble que la de las mujeres blancas en los Estados Unidos. Sin embargo, si bien se han descubierto más de 75 loci genéticos para la diabetes tipo 2 en poblaciones de ascendencia europea, asiática y mexicana, solo se han descubierto 3 variantes nuevas en estudios de asociación de todo el genoma (GWAS) de poblaciones de ascendencia africana; la más notable es SNP TCF7L2 rs7903146, la variante más significativamente asociada con la diabetes tipo 2 hasta la fecha.
TCF7L2 es un factor de transcripción y un componente clave de la vía de señalización Wnt (del que ya hemos hablado anteriormente), y está involucrado en el desarrollo de una amplia variedad de linajes y órganos celulares. Los mecanismos potenciales a través de los cuales las variantes de TCF7L2 influyen en la DM2 incluyen su papel en la adipogénesis, la miogénesis y el desarrollo de islotes pancreáticos, así como en la supervivencia de las células beta y la función del gránulo secretor de insulina. La proteína TCF7L2 aumenta durante la adipogénesis en las células 3T3-L1 y las células madre primarias de adipocitos. La inactivación de la proteína TCF7L2 mediante la eliminación del dominio de unión al ADN del grupo de alta movilidad (HMG), en los adipocitos maduros in vivo, conduce a intolerancia a la glucosa en todo el cuerpo y resistencia a la insulina hepática. Este fenotipo está asociado con una mayor masa de tejido adiposo subcutáneo, hipertrofia de adipocitos e inflamación. También participa en la regulación transcripcional de los genes para el proglucagón y de los péptidos similares al glucagón GLP-1 y GLP-2, que juegan un papel transcendente en la secreción de insulina posprandial.
Los polimorfismos TCF7L2 se han asociado con una secreción alterada de insulina, a través de efectos directos sobre las células beta de los islotes pancreáticos. De hecho, la desregulación del metabolismo de la glucosa, la disminución del procesamiento de la proinsulina y los niveles elevados de GIP y HbA1c se observan incluso en individuos normoglucémicos, con polimorfismos TCF7L2, antes del inicio de la DM2.
Figura 7: Incidencia de diabetes (casos / 100 años-persona) según el genotipo
en rs7903146 y el brazo de tratamiento en el Programa de prevención de diabetes.
Florez JC, Jablonski KA, Bayley N, et al. TCF7L2 polymorphisms and progression
to diabetes in the Diabetes Prevention Program. N Engl J Med 2006;355:241–250.
| Por otra parte, hay evidencia de la interacción entre las variantes TCF7L2 y los espectros de ácidos grasos libres y por lo tanto de la implicación del gen en el metabolismo de los lípidos. Sin embargo, se necesitan más estudios para dilucidar la mecanismos por los cuales el haplotipo de riesgo TCF7L2 realmente influye en la proporción de ácidos grasos poliinsaturados en ayunas, de ácidos grasos monoinsaturados y de ácidos grasos saturados (14). |
- La variante rs7903146
Se ha confirmado una asociación muy significativa entre la susceptibilidad relacionada con la DM2 y las variantes comunes en el TCF7L2 en sujetos islandeses, y el resultado fue el mismo con el método de caso-control en cohortes danesas y estadounidenses. El alelo T del SNP rs7903146 aumentó el riesgo de DMT2 en la población francesa con 2367 casos y 2499 controles. Los mismos resultados fueron publicados en el caso de población japonesa, afroamericana y brasileña. Sin embargo, no se encontró asociación entre rs7903146 y DM2 en población china.
En cualquier caso, la calidad de los datos varía mucho en diferentes ensayos, lo que se considera una de las razones de los resultados inconsistentes, además de los pequeños tamaños de muestra. Para mejorar los resultados estadísticos se ha utilizado la estrategia de metaanálisis combinando muestras. En 2018 Weiyue Ding et al. realizaron un metanálisis de estudios publicados que incluían rs7903146 en relación con DM2 con el objetivo de lograr un resultado más completo. Se utilizaron un total de 28 estudios para reevaluar la asociación entre rs7903146 y DM2.
En total se incluyeron 56628 participantes (34232 casos y 22396 controles) de 28 estudios recogidos en 26 artículos de la literatura. El resultado de cuatro subgrupos (caucásico, asiático oriental, asiático meridional y otros) sugirió que rs7903146 se asocia significativamente con DM2 en todos los subgrupos y grupos valorados (15, 16, 17, 18, 19).
- Implicaciones clínicas
También se explora la posibilidad de que las variantes TCF7L2 pudieran servir como marcadores para adaptar regímenes terapéuticos personalizados. Por ejemplo, DPP4 es una peptidasa que degrada las incretinas como GLP-1. Los inhibidores DPP4 mejoran la secreción de insulina estimulada por incretina e inhiben la liberación de glucagón, lo que ayuda a normalizar los niveles de glucosa en sangre. Si se confirma una influencia negativa en la secreción de incretina como un eje patológico principal en la diabetes tipo 2 impulsada por expresion de variantes TCF7L2, los inhibidores de DPP4 podrían servir como contrapeso y revertir los efectos de las variantes de TCF7L2 en los niveles de incretina. Los polimorfismos TCF7L2 se han asociado con variaciones en la respuesta al tratamiento con sulfonilurea, pero estos efectos son probablemente demasiado modestos para dirigir el tratamiento.
Sin embargo, hay que hacer algunas consideraciones sobre la utilidad clínica de las variantes TCF7L2. En primer lugar, las variantes específicas de riesgo de DM2 de TCF7L2 varían de una población a otra. Se han observado tamaños de efectos similares en varios grupos étnicos para las variantes TCF7L2 más estudiadas, pero la investigación debe ampliarse más allá de las poblaciones no blancas. En segundo lugar, aunque los tamaños del efecto de las variantes de TCF7L2 son relativamente grandes, una imagen mucho más precisa de la estratificación del riesgo y la personalización terapéutica se basaría en una colección más amplia de genes de predisposición en lugar de TCF7L2 solo.
En cualquier caso, TCF7L2 podría ser un biomarcador prometedor para la prevención de enfermedades para aquellos sujetos con una elevada predisposición genética a la DM2. Pero no existe un papel clínico para el cribado de rutina para TCF7L2, y la presencia de variantes de TCF7L2 que están asociadas con un mayor riesgo de DM2, sino que debería considerarse en combinación con otros predictores conocidos, al evaluar el riesgo individual de un paciente para DM2 (20, 21, 22).
7. Epistasis
Epistasis es la interacción gen-gen para expresar un determinado fenotipo. La expresión de un gen se ve modificada por la acción de uno o varios otros genes. Al gen cuyo fenotipo se está expresando se le llama epistático, mientras que al fenotipo alterado o suprimido se le llama hipostático. Este fenómeno puede darse tanto entre genes que segreguen de forma independiente como entre loci que estén ligados; si bien, en el caso de genes ligados variarán las frecuencias fenotípicas esperadas en la descendencia debido a los efectos de la recombinación.
La interacción gen-gen también puede afectar al resultado de los estudios de asociación gen-enfermedad. Las interacciones epistáticas pueden ser de proteínas-proteínas, proteínas-genes, interacciones ARN-proteínas o silenciamiento de ARN. Genes, ARN, proteínas y enzimas suelen formar parte de una o varias vías, muchas de las cuales están interconectadas. El concepto de amortiguación bioquímica u homeostasis, explica el proceso: los sistemas biológicos intentan mantener el equilibrio.
Las interacciones gen-gen pueden ser específicas de alelos y pueden ser aditivas, negativas o multiplicativas. Por ejemplo: los sujetos que portan el alelo G de adiponectina y el alelo PPARγ2 ProAla12 parecen más sensibles a la insulina que aquellos que codifican el alelo T de adiponectina, las interacciones entre los genotipos adiponectina y PPARγ2 también contribuyen a las concentraciones de insulina en ayunas, las concentraciones de insulina en las pruebas de tolerancia a la glucosa oral y en el índice de resistencia a la insulina. Por tanto, las interacciones con genotipos PPARγ2 modifican la asociación de la adiponectina con la sensibilidad a la insulina (23).
Las interacciones epistáticas se producen debido a variantes genéticas que un individuo hereda. La frecuencia de los alelos difiere entre poblaciones ancestrales, de modo que la probabilidad de heredar variantes genéticas causales o interactivas varía. La deriva genética, las pequeñas poblaciones existentes durante períodos prolongados o la presión selectiva, influyen en la selección de alelos. Pequeñas diferencias en las frecuencias alélicas entre las poblaciones y, por lo tanto, entre los individuos, pueden conducir a diferencias en las respuestas biológicas, incluyendo respuestas a la dieta.
Las interacciones gen-gen pueden ser específicas de alelos y pueden ser aditivas, negativas o multiplicativas. Por ejemplo: los sujetos que portan el alelo G de adiponectina y el alelo PPARγ2 ProAla12 parecen más sensibles a la insulina que aquellos que codifican el alelo T de adiponectina, las interacciones entre los genotipos adiponectina y PPARγ2 también contribuyen a las concentraciones de insulina en ayunas, las concentraciones de insulina en las pruebas de tolerancia a la glucosa oral y en el índice de resistencia a la insulina. Por tanto, las interacciones con genotipos PPARγ2 modifican la asociación de la adiponectina con la sensibilidad a la insulina (23).
Las interacciones epistáticas se producen debido a variantes genéticas que un individuo hereda. La frecuencia de los alelos difiere entre poblaciones ancestrales, de modo que la probabilidad de heredar variantes genéticas causales o interactivas varía. La deriva genética, las pequeñas poblaciones existentes durante períodos prolongados o la presión selectiva, influyen en la selección de alelos. Pequeñas diferencias en las frecuencias alélicas entre las poblaciones y, por lo tanto, entre los individuos, pueden conducir a diferencias en las respuestas biológicas, incluyendo respuestas a la dieta.
8. Epigenética
La epigenética estudia los mecanismos de expresión de los genes sin modificación de la secuencia de ADN de los mismos. Es decir, se refiere a cambios heredables, que abarcan un conjunto de reacciones químicas y otros procesos, capaces de modificar la actividad del ADN y, por tanto, la función de los genes, sin alterar la secuencia de nucleótidos que lo componen. Son conocidos en el proceso epigenético los mecanismos de metilación de bases, la modificación postraduccional de histonas (por procesos de acetilación, metilación, deaminación, isomerización de prolinas y ubiquitinización) y el ARN no codificante o ARN de interferencia (iARN), a los que nos referimos a continuación.
 |
| Figura 8: Mecanismos epigenéticos |
- Metilación de ADN
La metilación de ADN consiste en la unión de un grupo metilo a la posición 5' de los residuos de citosina en un dinucleótido Citosina-fosfato-Guanina (CpG) (*) por las metiltransferasas de ADN (DNMT, incluidos DNMT1, DNMT3A y DNMT3B). Además de los DNMT, las desmetilasas de la familia de translocación 10-11 (TET) de las dioxigenasas de ADN (TET1/2/3) también controlan el estado de metilación del genoma, al eliminar el grupo metilo de las citosinas metiladas. La S-adenosilmetionina (SAM) es el donante del grupo metilo en las reacciones catalizadas por DNMT, mientras que Fe(II)-, α-cetoglutarato-(α-KG), ascorbato y O2 son los cofactores de la oxidación catalizada por TET de 5-metilcitosina (5mC) a 5-hidroximetilcitosina (5hmC). A diferencia de la metilación, la desmetilación del ADN generalmente activa la transcripción.
La adición de un grupo metilo a la base citosina permite la conformación cerrada de la cromatina. Ello significa que una elevada tasa de metilación se asocia con el silenciamiento de los genes. El grado de metilación se relaciona con diversos factores externos al genoma, entre los que se encuentran los factores ambientales. En mamíferos, algunas sustancias provenientes de la dieta tienen la capacidad de añadir grupos metilos, por ejemplo aminoácidos como la metionina (HO2CCH(NH2)CH2CH2SCH3); o nutrientes esenciales como la colina (serie de sales cuaternarias de amonio que contienen el catión N,N,N- trimetiletanolamina, que suelen agrupar con vitaminas del grupo B); vitaminas como el ácido fólico (vitamina B9) y la piridoxina (vitamina B6). Son sustancias que se encuentran habitualmente en la dieta. La reacción de metilación precisa de la intervención de la enzima ADN-metiltransferasa. La metilación es un proceso dinámico y la actividad de la enzima es la encargada de mantener los patrones de metilación. Las marcas de metilación están relacionadas con las proteínas de unión metil-CpG (*).
(*) Las islas CpG de vertebrados (CGI) son secuencias cortas de ADN intercaladas, que se desvían significativamente del patrón genómico promedio, al ser ricas en GC (Guanina y Citosina) y predominantemente no metiladas. La mayoría, quizás todos, los CGI son sitios de iniciación de la transcripción. Los CGI están genéricamente equipados para influir en la estructura local de la cromatina y simplificar la regulación de la actividad genética. Los CGI son distintos en los vertebrados debido a su falta de metilación del ADN y la ausencia de deficiencia de CpG.
Un creciente cuerpo de evidencia sugiere que muchas manifestaciones del envejecimiento son epigenéticas. Un tipo particular de control epigenético: la metilación de la citosina-5´ dentro de los dinucleótidos CpG se relaciona con el proceso de envejecimiento. Existe evidencia de desregulación epigenética por alteraciones de la metilación relacionadas con la edad (24, 25, 26, 27).
- Modificación de histonas
Las histonas son proteínas básicas, de bajo peso molecular. Forman la cromatina, junto con el ADN, sobre la base, entre otras, de unas unidades conocidas como nucleosomas (estructura sobre la que se enrolla el ADN). Por modificaciones post-trasduccionales (procesos de acetilación, fosforilación, metilación, deaminación, isomerización de prolinas y ubiquitinización) se puede modificar la configuración de las histonas. Combinaciones específicas en la modificación de las histonas sirven como una especie de código que determina si el gen ha de ser silenciado o expresado y esta es otra forma de regulación génica.
La modificación mas frecuente e importante de las histonas es la acetilación, que se produce en presencia de acetil Co-A, mediante la enzima acetiltransferasa de la histona. Mientras que la enzima histona desacetilasa es la encargada de eliminar la marca epigenética.
La acetilación de histonas interviene en la regulación transcripcional en las células normales: En el caso de histonas desacetiladas la cromatina presenta una conformación cerrada y, por tanto, los factores de transcripción no pueden acceder al DNA, lo que inhibe la expresión génica. Por el contrario, en el caso de las histonas acetiladas, la cromatina presenta una conformación abierta y, por tanto, los factores de transcripción pueden acceder al DNA, lo que favorece la expresión génica. Los primeros estudios bioquímicos de cromatina CGI aislada mostraron altos niveles de acetilación de histonas H3 y H4, que son características de la cromatina transcripcionalmente activa.
Las histonas también pueden ser modificadas por metilación, que tiene lugar sobre resíduos de arginina y especialmente de lisina. En ello interviene la enzima lisina metil transferasa, lo que da lugar a S-adenosilhomocisteina. La lisina desmetilasa elimina específicamente la metilación de las histonas.
- ARN no codificante (ncARN)
El ácido ribonucleico (ARN o RNA) es un ácido nucleico formado por una cadena de ribonucleótidos (nucleótidos del ARN formados por una molécula de fosfato, el azúcar ribosa y una base que puede ser: uracilo, la adenina, la citosina o la guanina). Está presente tanto en las células procariotas como en las eucariotas, y es el único material genético de ciertos virus (virus ARN).
 |
| Figura 9: ARN mensajero |
El ARN mensajero (ARNm) es la molécula que dirige las etapas intermedias de la síntesis proteica. El ARNm transfiere la información del ADN a los ribosomas para la síntesis de proteínas. Otros tipos de ARN son el ARN de transferencia (ARNt) (actúa durante la traducción transfiriendo un aminoácido a la molécula polipeptídica en formación en el ribosoma); ARN ribosomal (ARNr) representa los 2/3 de la estructura ribosómica, combinado con proteínas, y el 80% del ARN citoplásmico en eucariotas.
El ncARN es otra forma de regulación génica, que tiene lugar mediante la acción del llamado ARN de interferencia (iARN). El iARN no codifica para una proteína específica, pero impide la traducción del ARNm. Se trata de una regulación negativa de la expresión a nivel post-transcripcional: silenciamiento génico post-transcripcional.
Los exones de genes codificadores de proteínas conocidos representan menos del 3% del genoma humano. El 97% restante es territorio en gran parte inexplorado, con solo una pequeña fracción caracterizada. Del genoma total que se transcribe, solo el 2% codifica proteínas, mientras que la gran mayoría se transcribe como ARN no codificante.
El genoma que no codifica proteínas está ganando importancia en biología y medicina. Los ncARN más estudiados son los microARN (miARN), pero muchos otros tipos de ncARN también tienen un papel importante en la homeostasis y la enfermedad celular: siARN (small interfering ARN) y piARN (Piwi interacting ARN), ARN antisentido, pequeños ARN nucleolares (snoARN), ARN largo no codificante (ARNnc largo o long ncARN o lncARN), grandes ARN intergénicos no codificantes (lincARN) y transcritos de regiones ultraconservadas (T-UCR).
 |
Figura 10: Mecanismo de RNAi/ribointerferencia
mediado por siRNA. Mocellin, S., Provenzano, M.
RNA interference: learning gene knock-down
from cell physiology. J Transl Med 2004; 2:39.
|
La lista no es exclusiva, y en muchos casos no existe una distinción molecular clara entre las clases, en parte porque muchos ncRNA tienen funciones desconocidas. Sin embargo, hay algunos lncRNA bien estudiados, como el ARN antisentido de transcripción homeobox (HOX) (HOTAIR) y el transcrito específico de inactivación X (XIST), que están involucrados en la regulación transcripcional.
Las diferencias conocidas entre ellos se basan en el tamaño, en la estructura de su cadena (siARN es bicatenario, mientras que miARN y piARN son monocatenarios) y, por su puesto en sus mecanismos de interferencia. Los dos primeros (siARN y miARN) son dependientes de la enzima Dicer, que cataliza el primer paso de formación de iARN.
Un modelo de mecanismo de interferencia de ARN sería, por ejemplo, el siguiente: La aparición de ARN bicatenario (ds) dentro de una célula (por ejemplo, como consecuencia de infección viral) desencadena una respuesta compleja, que incluye entre otros fenómenos (por ejemplo, producción de interferón y sus consecuencias) una cascada de eventos moleculares conocidos como ARNi. Durante este proceso, la enzima celular Dicer se une al dsARN y lo divide en piezas cortas de aproximadamente 20 pares de nucleótidos de longitud conocida como siARN. Estos pares de ARN se unen a la enzima celular llamada complejo silenciador inducido por ARN (RISC) que usa una cadena del piARN para unirse a una cadena simple de ARNm, de secuencia complementaria. La actividad nucleasa de RISC luego degrada el ARNm, silenciando así la expresión del gen viral. Del mismo modo, se cree que la maquinaria genética de las células utiliza iARN para controlar la expresión de ARNm endógeno, agregando así una nueva capa de regulación postranscipcional (Figura 9).
Los piARN (ARN que interactúan con PIWI) (*) son cadenas de 29-30 nucleótidos. Se generan a partir de precursores largos monocatenarios. Su formación es un proceso independiente de la proteína Dicer y se asocian a una proteína del tipo Argonauta, concretamente AGO2.
(*) PIWI es una de las dos subfamilias de proteínas tipo Argonauta (Figura 11). Estas proteínas son endonucleasas catalíticas en el complejo de silenciamiento inducido por ARN, el complejo proteico que media el fenómeno de interferencia de ARN. Las proteínas Piwi tienen actividad "slicer" o cortadora, conferida por uno de los dominios de la estructura de la proteína. Las proteínas Piwi están altamente conservadas como proteínas de unión a ARN y se encuentran presentes tanto en eucariotas como arqueas.
Endonucleasas catalíticas en el complejo de
silenciamiento inducido por ARN de interferencia.
Un ARN antisentido es la hebra complementaria (no codificadora) de una hebra de ARNm (codificadora). La mayoría de los ARN antisentido inhiben genes, pero unos pocos activan la transcripción.
Los lncARN, que se definen como transcripciones que no codifican proteínas de más de 200 nucleótidos, regulan en muchos circunstancias la expresión génica en eucariotas. En determinadas poblaciones celulares, una cuarta parte de los genes que codifican para proteínas y el 80% de los lncARN detectados en el genoma humano, están presentes en una o ninguna copia por célula, ya que existe una restricción en determinados ARN.
Los LncARN se consideran reguladores fundamentales de la estructura del genoma y la expresión génica. Pueden interactuar directamente con las enzimas modificadoras de la cromatina y los factores de remodelación de nucleosomas, para controlar la estructura de la cromatina y la accesibilidad a la información genética. Además, la expresión de lncARN puede controlarse por factores de remodelación de la cromatina, lo que sugiere un circuito de regulación de retroalimentación. Se les considera importantes reguladores de la fisiología y patología celular. Sin embargo, la mayoría de los lncARN siguen siendo desconocidos por sus funciones biológicas, su papel en la enfermedad y los mecanismos precisos que protagonizan.
Se ha revelado una amplia asociación de lncARN con la maquinaria epigenética para controlar la estructura de la cromatina y la expresión génica. Los LncARN pueden interactuar directamente con muchas enzimas modificadoras de histonas y ADN para participar en modificaciones covalentes de histonas o ADN. Además, se ha puesto de manifiesto que un lncARN es capaz de modular el proceso de remodelación de la cromatina no covalente y dependiente de ATP.
Efectivamente, se ha identificado un grupo de lncARN asociados con el gen Myh7 de la cadena pesada de miosina en ratones. El transcrito de ARN asociado a la cadena pesada de miosina se denominó Myheart (Mhrt). Mhrt puede proteger al corazón de la hipertrofia e insuficiencia cardíaca inducida por el estrés, a través de inhibición directa de la función de la cromatina Brg1. Brg1 es una subunidad catalítica ATPasa del complejo de remodelación de cromatina BAF (*) similar a SWI/SNF, y es esencial para el desarrollo de la hipertrofia cardíaca patológica.
(*) Cuando una célula necesita adaptarse (por ejemplo, para responder a señales de desarrollo o ambientales o a daños en el ADN), necesita alterar la accesibilidad de su ADN. Hacer exactamente esto es la función de los remodeladores de cromatina, enzimas que usan la energía del ATP para mover o desalojar nucleosomas. Los complejos remodeladores de cromatina son diferentes en las células humanas: un complejo particularmente interesante es el complejo BAF. De hecho, no se trata de un solo complejo, sino de muchos diferentes. Hasta 15 posiciones complejas pueden ser ocupadas por proteínas codificadas de 29 genes diferentes, los combinatorios suman más de 10.000 complejos diferentes teóricamente posibles.
Los lncARN pueden actuar a través de 2 mecanismos diferentes para modular de forma covalente la cromatina. Primero, los lncARN pueden unirse directamente a las enzimas modificadoras de cromatina, sirviendo así como una guía para anclar los modificadores de cromatina a regiones genómicas específicas o funcionar como señuelo para secuestrar modificadores de cromatina de sitios genómicos específicos. En segundo lugar, los lncARN pueden incorporarse al complejo modificador de la cromatina y funcionar como parte del complejo o como un andamiaje para ensamblar el complejo para la modificación de la cromatina. (Figura 12) (28, 29, 30, 31, 32).
Los exones de genes codificadores de proteínas conocidos componen menos del 3% del genoma humano. El 97% restante es territorio en gran parte inexplorado, con solo una pequeña fracción caracterizada. La detección de transcripción en este territorio intergénico ha mostrado que las regiones intergénicas codifican ARN no codificantes de mayor longitud (lincARN) que los descritos previamente. Se han identificado decenas de miles de lincARN. Estos lincRNAs están específicamente regulados y conservados, demostrándose que no son producto del “ruido” transcripcional. Además, los lincRNA están fuertemente enriquecidos para los SNP asociados a rasgos que sugieren un nuevo mecanismo por el cual las regiones asociadas a rasgos intergénicos pueden ser funcionantes (33).
Los factores ambientales son fuente de modificaciones importantes en los genes. La regulación epigenética es responsable de los mecanismos adaptativos de los seres vivos. La plasticidad del genoma le concede la capacidad para ser modificado y modulado y el resultado es la formación de diferentes fenotipos, que responden a los diferentes factores ambientales. Estos cambios epigenéticos tienen un elevado grado de estabilidad y pueden ser heredables, manteniéndose activos en una línea celular durante varias generaciones. Pueden generarse fenotipos beneficiosos para la supervivencia, pero también se pueden producir errores en las modificaciones inducidas por el ambiente, que pueden heredarse y perdurar. Estos “errores” constituyen, en muchos casos condiciones para la aparición de fenotipos patológicos (34).
Dentro de la estructura molecular cromosómica existen tres niveles o capas: genes codificadores de proteínas, genes no codificadores y capa epigenética. El nivel epigenético resulta crucial para el desarrollo. En él se encuentran las claves del crecimiento, del envejecimiento y del desarrollo del cáncer y de otras enfermedades muy prevalentes. Los procesos epigéneticos, como ya hemos señalado, no cambian la secuencia de nucleótidos del ADN, pero influyen en la expresión del mismo.
El riesgo de desarrollar DM2 está muy influenciado por factores ambientales. Dado que nuestro código genético no cambia significativamente en una o dos generaciones, el incremento constante de prevalencia de la diabetes se debe principalmente a los cambios en el medio ambiente. Las epidemias de obesidad y DM2 son paralelas entre sí y la creciente prevalencia de ambas está relacionada con los cambios en los hábitos alimenticios y en el estilo de vida.
Los genes influyen de múltiples maneras en el riesgo de desarrollar DM2, pero no solo al alterar la secreción de insulina o la acción de la misma. También influyen sobre la forma en que cualquier individuo interactúa con los factores ambientales. La adopción de estilos de vida poco saludables y la disposición a cambiar dichos estilos de vida varía entre individuos de una misma población. La genética también influye en factores como el gusto y las preferencias alimentarias; así como sobre el mayor o menor gasto energético en reposo o el tipo de microbioma de cada individuo y, lo que es muy importante en la DM2, sobre su relación con el estilo de vida. Estas interacciones genético-ambientales pueden ser extremadamente complejas y son una de las razones por las que solo una pequeña proporción de la herencia de DM2 se ha podido explicar hasta la fecha.
Los avances en genética humana ha llevado a la identificación de un número relativamente grande de loci asociados a obesidad y DM2. Sin embargo, su contribución al riesgo de enfermedad es pequeña y su valor predictivo bajo, lo que sugiere que el estilo de vida juega un papel crucial en la obesidad y el desarrollo de DM2. Los estudios que investigan las interacciones entre el gen y el estilo de vida en la obesidad y la DM2 han sugerido que el efecto biológico de la predisposición genética puede ser parcial o totalmente abolido por la modificación del estilo de vida o el estilo de vida saludable y viceversa.
La importancia de los factores ambientales y de estilo de vida se ha puesto de manifiesto mediante estudios epidemiológicos, que han mostrado cómo el efecto negativo de varios genes, asociados con la obesidad y la DM2, puede atenuarse en individuos con niveles más altos de actividad física o estilos de vida saludables. Por el contrario, un patrón bajo de actividad física y dieta occidental (no en el caso de la dieta mediterránea) lo acentúa. Además, se ha descubierto que la actividad física, así como las intervenciones dietéticas y de estilo de vida combinadas, inducen una significativa disminución del peso corporal y de otros rasgos antropométricos, contribuyendo a abolir el riesgo de obesidad en portadores de alelos de riesgo de dos de los genes más fuertemente asociados con la obesidad: FTO y MC4R.
Con respecto a los resultados de DM2 en estudios a gran escala, hay pruebas sólidas de la mejora de los rasgos metabólicos y la atenuación del riesgo de diabetes entre los portadores de alelos de riesgo TCF7L2 mediante dieta y ejercicio. El alelo menor del gen PPARγ también se ha asociado con un riesgo sustancialmente mayor de DM2 y aterosclerosis. Sin embargo, se ha sugerido que la modificación del estilo de vida atenúa su efecto negativo sobre el perfil metabólico, el peso corporal y el riesgo de diabetes (36, 37).
Cambios durante la gestación, en el ambiente intrauterino materno, y en la infancia temprana, pueden alterar el riesgo de enfermedades crónicas para toda la vida. Por ejemplo, los bebés que nacen pequeños para la edad gestacional tienen un mayor riesgo de desarrollar obesidad y DM2 en la edad adulta. La mayor parte de este riesgo puede deberse a cambios epigenéticos en genes críticos. Ensayos en animales y estudios iniciales en humanos sugieren que tales mecanismos pueden explicar el impacto de la nutrición intrauterina y el peso al nacer sobre el riesgo futuro de diabetes, obesidad y síndrome metabólico. Por tanto, la herencia DM2 puede deberse, al menos en parte, a cambios epigenéticos en la vida intrauterina, resultado de influencias ambientales maternas, en lugar de a variaciones heredadas en la secuencia de ADN (21).
En la DM2 la hiperglucemia causa inflamación sistémica de bajo grado por diferentes vías, incluida la producción de ROS (reactive oxygen species) y AGE (advanced glycation end products) y la sobreexpresión de TLR-4. La inflamación, a su vez, favorece el desarrollo de hiperglucemia al actuar tanto en las células β pancreáticas (donde regula negativamente la síntesis de insulina, la secreción y la biología celular general) como en los tejidos dirigidos a la insulina, es decir, hígado, músculo esquelético y adiposo. tejido (donde promueve la resistencia a la insulina). Se establece así un circulo vicioso lesional (Figura 14).
La epigenética abarca un grupo de mecanismos de control genómico resultante de los cambios en la estructura de la cromatina sin alteraciones en la secuencia de ADN. Estos mecanismos median la respuesta genómica a los estímulos externos mediante la regulación del silenciamiento o activación de genes. Las alteraciones en estos mecanismos pueden conducir a disfunciones patológicas. El patrón alterado de expresión génica, que subyace en la resistencia progresiva a la insulina y así como su deficit de secreción en la DM2 dependen de cambiós epigenéticos en sus tres diferentes formas: metilación de ADN, modificación postraduccional de histonas y relativos al ARNnc, a los que ya nos hemos referido (38).
Durante el desarrollo, genes inapropiados para un determinado tipo de célula se metilan en las islas CpG, reprimiendo de manera estable y hereditaria su expresión. Por ejemplo, la metilación de islas CpG (CGI) en el gen de la insulina, en células no productoras de insulina, impide su expresión inapropiada, mientras que el promotor de insulina es desmetilado apropiadamente en la célula β pancreática. Igualmente, para mantener la identidad y función de las células β, ciertos genes esenciales deben permanecer metilados, mientras que los genes perjudiciales permanecen metilados y suprimidos.
En la DM2 la hiperglucemia causa inflamación sistémica de bajo grado por diferentes vías, incluida la producción de ROS (reactive oxygen species) y AGE (advanced glycation end products) y la sobreexpresión de TLR-4. La inflamación, a su vez, favorece el desarrollo de hiperglucemia al actuar tanto en las células β pancreáticas (donde regula negativamente la síntesis de insulina, la secreción y la biología celular general) como en los tejidos dirigidos a la insulina, es decir, hígado, músculo esquelético y adiposo. tejido (donde promueve la resistencia a la insulina). Se establece así un circulo vicioso lesional (Figura 14).
La epigenética abarca un grupo de mecanismos de control genómico resultante de los cambios en la estructura de la cromatina sin alteraciones en la secuencia de ADN. Estos mecanismos median la respuesta genómica a los estímulos externos mediante la regulación del silenciamiento o activación de genes. Las alteraciones en estos mecanismos pueden conducir a disfunciones patológicas. El patrón alterado de expresión génica, que subyace en la resistencia progresiva a la insulina y así como su deficit de secreción en la DM2 dependen de cambiós epigenéticos en sus tres diferentes formas: metilación de ADN, modificación postraduccional de histonas y relativos al ARNnc, a los que ya nos hemos referido (38).
Durante el desarrollo, genes inapropiados para un determinado tipo de célula se metilan en las islas CpG, reprimiendo de manera estable y hereditaria su expresión. Por ejemplo, la metilación de islas CpG (CGI) en el gen de la insulina, en células no productoras de insulina, impide su expresión inapropiada, mientras que el promotor de insulina es desmetilado apropiadamente en la célula β pancreática. Igualmente, para mantener la identidad y función de las células β, ciertos genes esenciales deben permanecer metilados, mientras que los genes perjudiciales permanecen metilados y suprimidos.
- Alteraciones en los patrones de metilación del ADN pueden tener importantes efectos metabólicos. Por ejemplo, la falta de metilación materna en la región cromosómica 6q24 da como resultado una forma rara de diabetes neonatal transitoria (TND). En este trastorno, la diabetes relacionada con la disfunción de las células β se produce a los pocos días del nacimiento, pero remite durante la infancia, mientras que los afectados suelen desarrollar DM2 más adelante. La región 6q24 del genoma contiene dos productos: una transcripción no traducida, de la que se ignora el significado funcional, como el gen para el adenoma pleomórfico 1 (PLAG1 ó ZAC). PLAG1 es un factor de transcripción de zinc finger o dedo de zinc (ZF) (*), candidato principal para TND, relacionado con la regulación del ciclo celular y con los fenómenos de apostosis. En la célula β, este gen también se ha relacionado con una secreción de insulina alterada. Por otra parte, es interesante considerar que en la comunidad afroamericana el cromosoma 6q24 está asociado con la obesidad y la DM2 en adultos. Sigue en discusión si esta relación se debe a un fenómeno epigenético. Lo que si parece claro es que los cambios heredables tras la metilación de ADN son relevantes para el desarrollo de la enfermedad metabólica.
(*) Los ZF (zinc finger) son motivos estructurales presentes en gran cantidad de proteínas eucariotas. Pertenecen al grupo de las metaloproteínas, capaces de unir el ion metálico zinc, que requieren para su funcionamiento. Se predice que más de 1500 dominios ZF existen en alrededor de 1000 proteínas diferentes en humanos. Las proteínas con motivos ZF son de las más abundantes en el genoma de organismos eucariotas y participan en diversidad de procesos celulares esenciales, entre los que destacan la transcripción genética, la traducción de proteínas, metabolismo, el plegamiento y ensamblaje de otras proteínas y de lípidos, muerte celular programada, entre otros.
La investigación sobre el papel de la metilación del ADN, en el desarrollo de la diabetes, reveló cambios significativos en los patrones generales de metilación en los islotes de donantes con DM2 en comparación con los islotes de los controles no diabéticos. Los grupos de proteínas que mostraron metilación alterada incluyeron genes relacionados con tres amplias categorías: vías de señalización esenciales para la adaptación de las células β; vías relacionadas con la supervivencia o, por el contrario, con la apostosis de la célula β y otras vías de significado incierto en relación con la célula β (39).
- Metilación diferencial del ADN en islotes de Langerhan en DM2
Los estudios de los perfiles de metilación de ADN diabético demostraron que estos son más similares entre sí que con el perfil de metilación de cualquier muestra de control. Ello indica la posibilidad de un perfil de metilación de ADN específico de DM2. Por otra parte, en estos estudios, los cambios de metilación del ADN son suficientemente pronunciados (incluso en los conjuntos de datos sin filtrar) para distinguir las muestras de diabetes de las de control.
El análisis de los componentes principales del conjunto de datos y la comparación de los mismos con el estado de DM2, mediante correlación biserial, permitió detectar cambios distintivos, específicos de DM2, en el epigenóma de los islotes pancreáticos.
En un estudio exhaustivo de datos de metilación de ADN en islotes de DM2 se identificaron 276 sitios CpG, afiliados a 254 promotores de genes, que muestran metilación diferencial entre muestras normales y enfermas. De ellos, 266 CpG (el 96%), mostraron hipometilación frente a a solo el 4% de CpG hipermetilados. Este perfil de metilación es significativamente diferente de los que sucede, en general, en el cáncer, en que la hipometilación e hipermetilación específicas de los genes implicados se distribuye de forma mas o menos uniforme, aunque con relación a la metilación global del ADN los cánceres muestran predominio de hipometilación.
La metilación diferencial del ADN observada en los islotes DM2 puede ser causal o secundaria a la hiperglucemia inherente a la diabetes. Informes recientes han demostrado que la exposición transitoria de las células endoteliales aórticas a glucosa elevada induce cambios epigenéticos persistentes (metilación del ADN, monometilación H3K4, acetilación H3K9/H3K16). Sin embargo, el tratamiento con elevado contenido en glucosa no afectó significativamente a la metilación del ADN de 16 CpG en 13 promotores de genes probados.
Una pregunta importante con respecto a la metilación diferencial del ADN detectada en los islotes DM2 es si estos cambios epigenéticos son únicos para los islotes pancreáticos o si son parte de un patrón general que ocurre en diferentes tejidos. En este sentido se analizaron leucocitos de sangre periférica de pacientes con DM2 y se compararon sus perfiles de metilación de ADN con controles no diabéticos emparejados por edad e IMC. No se detectaron diferencias significativas en los niveles de metilación del ADN entre las muestras de sangre control y DM2 en ninguno de los loci de CpG analizados. Los resultados sugieren que el patrón de metilación observado en los islotes aparentemente no es un fenómeno general. Por otra parte, la sangre no es un tejido sustituto adecuado para estudiar los cambios epigenéticos relacionados con DM2 en los islotes pancreáticos. Es decir, el patrón de metilación relacionado con la DM2 parece una característica propia de los islotes enfermos.
La metilación diferencial del ADN puede correlacionarse con cambios en la expresión génica en un subconjunto de genes. La comparación de los datos de metilación y expresión sugiere que los cambios observados en los niveles de metilación del ADN del promotor no se limitan a genes silenciosos o de baja expresión, sino que también se producen en promotores de genes expresados. En los islotes DM2, observamos hipometilación en los promotores de estos genes activos.
Los estudios señalan la participación de alteraciones epigenéticas en la DM2, que tienden a confirmar la contribución de los hábitos de estilo de vida para su desarrollo. Se han descrito cambios epigenéticos en DM2, específicamente un perfil de metilación de ADN alterado en islotes pancreáticos de pacientes con DM2, con predominio de la hipometilación en secuencias fuera de CGI. La metilación aberrante afecta a más de 250 genes, un subconjunto de los cuales también se expresa de manera diferencial. La desregulación de estos genes en la DM2 puede estar relacionada principalmente con la funcionalidad de las células β, la muerte celular y la adaptación al estrés metabólico (Figura 15).
Además, se han señalado genes que pertenecen a procesos biológicos cuya participación en DM2 aún no se comprende completamente, como la inflamación y los transportadores de iones/canales/sensores. Es posible que los cambios de metilación del ADN descubiertos pudieran ser, por una parte, indicativos de reacciones de las células de los islotes a la condición diabética y, por otra parte, podrían ser causales de DM2. Es decir, los cambios de metilación de ADN observados, podrían ser causa o consecuencia de la condición diabética. Y ese dilema es un desafío para la búsqueda de evidencia sobre los efectos primarios de la alteración de la metilación en DM2.
En general, a partir de los datos disponibles, la metilación modificada en los promotores de algunos genes identificados podría ser consecuente y representar reacciones al ambiente diabético. En otros genes, las aberraciones de metilación podrían desempeñar un papel causal, relacionando la disfunción de los islotes y la patogénesis de DM2. Se necesitarán estudios futuros a gran escala. que involucren múltiples etapas del desarrollo de DM2. para dilucidar el papel de los cambios epigenéticos en las diversas etapas de la patogénesis de DM2. Es imposible obtener biopsias pancreáticas repetidas, por lo que estos estudios deberán basarse en tejidos sustitutos que aún no se han validado. La disponibilidad del perfil de metilación de islotes humanos actualmente descrito permitirá la futura búsqueda y validación de tejidos sustitutos (40)
- Genes hipometilados/hipermetilados y activados en DM2
Un ejemplo destacado de gen que afecta la supervivencia celular, hipometilado y presumiblemente activado en islotes de donantes con DM2, es el gen CDK5R1. Este gen codifica la proteína subunidad reguladora de la quinasa 5 dependiente de ciclina 5 o p35, activador directo de la quinasa 5 dependiente de ciclina (CDK5). CDK5 se activa en la célula β por glucosa alta y la citokina proinflamatoria interleucina 1β. La activación de CDK5 conduce a una serie de efectos perjudiciales en la célula β, incluida la disminución de la transcripción del gen de la insulina y la disminución de la expresión del factor de transcripción pancreático y duodenal homeobox 1 (Pdx-1), una proteína que desempeña un papel central en el desarrollo de las células β, así como en el mantenimiento del fenotipo de células β maduras. La falta de Pdx-1 conduce a la agenesia pancreática en humanos y ratones, mientras que la haploinsuficiencia causa una forma de diabetes tipo MODY4. Además de su efecto sobre Pdx-1, la activación de CDK5 también conduce a un aumento del estrés del retículo endoplásmico, a través de alteraciones en la expresión génica de la ATPasa de calcio del retículo sarcoendoplasmático 2b (SERCA2b). Otros genes con un papel funcional en la supervivencia y en la apoptosis, que han demostrado metilación alterada de ADN en los islotes DM2, en comparación con los islotes sanos, incluyeron a CASP10, BCL2, PPP2R4 y GSTP1 (39,40).
Existen evidencias, cada vez mas firmes, de que la de-diferenciación de las células β juega un papel de importancia en la fisiopatología de la DM2. En un estudio en ratones se demostró que la ablación del gen FoxO1 (que integra la proliferación de células β con la función adaptativa de las mismas) dio como resultado hiperglucemia, con reducción de la masa de células β tras estrés fisiológico. El estudio de rastreo de linaje puso de manifiesto que la pérdida de masa de células β se debió a de-disferenciación pero no a la muerte de células β. Las células β de-disferenciadas se convirtieron en células tipo progenitor (*) que expresan Neurogenina3, Oct4, Nanog y L-Myc.
(*) Una célula progenitora es una célula que, como una célula madre, tiene una tendencia a diferenciarse en un tipo específico de célula, pero con menor potencialidad que una célula madre. La diferencia más importante entre las células madre y las células progenitoras es que las células madre pueden replicarse indefinidamente, mientras que las células progenitoras pueden dividirse solo un número limitado de veces. La controversia sobre la definición exacta se mantiene y el concepto sigue evolucionando. La mayoría de las células progenitoras se describen como oligopotentes. En este punto de vista, se pueden comparar con células madre adultas. Pero se dice que los progenitores se encuentran en una etapa adicional de la diferenciación celular. Están en “a medio camino" entre las células madre y las células totalmente diferenciadas. Células endodérmicas progenitoras, activadas para los fatores Pdx1 y Ptf1a, dan origen à células progenitoras pancreáticas para a formación de los ductos, ácinos y células endocrinas pancreáticas (Figura 16) (41).
En dicho estudio, se observó, además, que un subconjunto de células β, deficientes en FoxO1, adoptó el destino de las células α, lo que resultó en hiperglucagonemia. Los autores proponen que existe un predominio la de-diferenciación sobre la apostosis o la muerte de las células endocrinas, en la historia natural del deficit de células β que acompaña a la DM2. Como conclusión final sugieren que el tratamiento de la disfunción de las células β debería restaurar la diferenciación, en lugar de promover la replicación de las mismas. Ello representa una ventana abierta a futuros tratamientos capaces de revertir la de-diferenciación.
Los modelos en animales de experimentación sugieren un papel adicional para la de-diferenciación de células β en la fisiopatología de DM2, lo que aumenta la posibilidad de que las alteraciones en las rutas epigenéticas en el estado diabético puedan conducir a una pérdida de expresión de genes clave en la identidad de las células β. Se observado que Pax4 estaba hipermetilado y, por lo tanto, silenciado en islotes de donantes humanos con DM2.
De forma similar a Pdx-1, Pax4 es esencial en el desarrollo y mantenimiento de células β. La falta de Pax4 durante el desarrollo da lugar a MODY tipo 9 y las mutaciones en Pax4 también se han asociado con la susceptibilidad tanto a DM1 como DM2. Además, se requiere Pax4 para la proliferación de células β en el páncreas adulto y para la adaptación del mismo a la hiperglucemia crónica.
También se ha demostrado que la expresión exógena de Pax4 reprograma las células α adultas en un fenotipo de células β (Figura 17). En ratones, células embrionarias productoras de glucagón en el páncreas pueden regenerarse y convertirse en células tipo β productoras de insulina a través de la expresión constitutiva/ectópica del gen Pax4. Sin embargo, se desconoce si las células α en ratones adultos muestran la misma plasticidad. Muchos de los mecanismos subyacentes a dicha reprogramación siguen sin estar claros y plantean varias cuestiones de interés (42):
¿Pueden las células α, que han sido sometidas a señalización ambiental, maduración y envejecimiento, retener la capacidad de convertirse en células similares a β tras la expresión de Pax4?
¿Podría restaurarse la masa celular similar a β, a partir de células glucagón+, tras ablación de las células β?
¿La limitación del número de células precursoras podría restringir la regeneración de células similares a β en ratones adultos?
¿Cual es el mecanismo subyacente en la neogénesis de células similares a β a partir de células glucagón+?
Los resultados de estudios recientes proporcionan respuestas a algunas de estas preguntas. Por ejemplo, hay evidencia de que las células α adultas también pueden convertirse en células similares a β funcionales, a cualquier edad, tras la expresión de Pax4, y que posteriormente se activa un ciclo de neogénesis de células endocrinas/islotes.
Tras la conversión celular mediada por Pax4 de células tipo α a β, las células precursoras del revestimiento del conducto pancreático se movilizan continuamente, reexpresan el gen de desarrollo Ngn3 y adoptan sucesivamente un fenotipo glucagón+ y una identidad celular de tipo β, que responden a estímulos fisiológicos, a través de un mecanismo que implica el despertar de la transición epitelial a mesenquimal.
Es importante destacar que tales procesos de regeneración controlables permiten varios ciclos de reposición de una masa celular completa tipo β que contrarresta la diabetes inducida por toxinas.
Estos procesos pueden regenerar repetidamente toda la masa de células β y, por lo tanto, revertir varias rondas de diabetes inducida por toxinas, proporcionando perspectivas terapéuticas para estrategias regenerativas.
Tomados en conjunto, todos estos datos sugieren que la metilación aberrante puede desempeñar un papel en la pérdida del programa transcripcional de células β normales y contribuir a la pérdida de la identidad de las células β en condiciones fisiopatológicas (40, 43, 44).
- Histonas ¿Memoria de la célula?
La identidad de una célula se define por su patrón de expresión génica y silenciamiento, su memoria consiste en mantener ese patrón de expresión génica a través de la replicación del ADN, el ensamblaje de la cromatina y el plegamiento del ADN que acompaña a la mitosis. El modo en que el ADN se “empaqueta” constituye una estructura que permite contener y reducir la longitud del ADN. El nucleosoma constituye la partícula central de esta estructura de plegamiento. Comprende un octámero de histonas centrales (dos de H2A, H2B, H3 y H4) alrededor de las cuales se envuelven 146 pares de bases de ADN en vueltas superhelicales.
Cada vez parece mas claro que el nucleosoma tiene un papel adicional, quizás igualmente importante y conservado, a saber, la regulación de la expresión génica. Y es probable que el nucleosoma pueda transmitir información epigenética de una generación celular a la siguiente y actuar como el banco de memoria de la célula.
El almacenamiento de información reside principalmente en las colas amino-terminales de las cuatro histonas centrales. Las colas están expuestas en la superficie del nucleosoma, sujetas a una variedad de modificaciones postraduccionales, catalizadas por enzimas de aminoácidos seleccionados, que incluyen acetilación de lisina, metilación de lisina y arginina, fosforilación de serina y fijación del péptido pequeño ubiquitina (ver mas adelante).
En la Figura 18 los números de residuos se muestran para cada modificación. H3 lisina 9 puede ser tanto acetilado como metilado. Se muestran los dominios de la cola C-terminal de una molécula H2A y una molécula H2B (líneas discontinuas) con sitios de ubiquitinación en H2A lisina 119 (más común en mamíferos) y H2B lisina 123 (más común en levadura). Las modificaciones se muestran solo en una de las dos copias de las histonas H3 y H4 y solo se muestra una cola para H2A y H2B. Los sitios marcados con flechas verdes son susceptibles de corte por tripsina en nucleosomas intactos. El gráfico se ha construido con un compendio de datos de varios organismos estudiadios, algunos de los cuales pueden carecer de alguna de estas modificaciones en particular (por ejemplo, no hay H3meK9 en S. cerevisiae) (45).
- Modificaciones en histonas en DM. Respuesta inflamatoria.
Estudios experimentales han demostrado la influencia del ambiente intrauterino, por mecanismos epigenéticos, en el desarrollo de disfunción de células β. Es el caso de retraso del crecimiento intrauterino (intrauterin growth retardation o IUGR), en ratones, se pone en marcha un mecanismo epigenético mediado por una alteración en la acetilacion de histonas de nucleosomas relacionados con el gen Pdx1(*), que lleva a la represión de la expresión de dicho gen (Figura 19).
(*) PDX1es un gen que tiene un rol determinante regulador durante la formación del páncreas embrionario. La mutación de este gen suele producir importantes alteraciones en la formación del páncreas embrionario, las cuales pueden desencadenar serios desequilibrios en la función del páncreas post-natal. Su alteración suele acarrear problemas en la diferenciación de las células del páncreas exocrino y en la función de las células beta del páncreas endocrino.
La histona H3 está hiperacetilada en los promotores del factor de necrosis tumoral α (TNFα) y la enzima ciclooxigenasa (COX-2) asociada a la inflamación en monocitos aislados de pacientes con DM1 o DM2, lo que sugiere linfluencia potencial, de la actividad de las HAT y HDAC, en la expresión de genes proinflamatorios en monocitos de pacientes que padecen diabetes.
La influencia específica de los HDACi en el sistema inmunitario, en relación con DM1 y DM2, está en estudio y discusión. Los estudios in vitro, parecen sugerir que la hiperacetilación es mas una consecuencia de la hiperglucemia, o de otra alteración metabólica, que causa de diabetes. En cualquier caso, además del estado hiperglucémico, el uso de HDACi (por ejemplo la tricostatina A, utilizada en dosis altas en monocitos de diabéticos) da como resultado un incremento de la acetilación de histona y estimula la producción de la citoquina IL-1β y TNFα, a través de la activación de NFκB. Además, la actividad de NFκB se incrementó por sobrexpresión de HAT y TSA, revirtiendo la sobreexpresión de HDAC.
Los HDCAi, como TSA, tienen efectos antiinflamatorios a dosis relativamente bajas, suficientes para inducir hiperacetilación de histona, pero con las que no se observan efectos indeseables. Por otra parte, otro inhibidor de HDCA, el ITF2357, ha mostrado una reducción de la respuesta inflamatoria en mononucleares de sangre periférica, por descenso de liberación de TNFα, secreción de IL-1β y síntesis de interferon γ (IFNγ).
Aunque no está completamente desvelado el potencial de los HDACi, su posible uso también es prometedor con respecto al tratamiento de complicaciones diabéticas tardías, como la nefropatía y la retinopatía diabética (Figura 20) (47, 48, 49, 50, 51).
- Modificación de histonas y desarrollo de células β
El objetivo de la metilación de histonas no es solamente el de mantener el ADN empaquetado como heterocromatina inaccesible a factores de transcripción, sino que, dependiendo del resíduo de histona metilado, puede tener diferentes efectos sobre la transcripción de genes. En este sentido, por ejemplo, la dimetilación de Lys-4 de la histona H3, se asocia con una conformación abierta de eucromatina. La metilación de los residuos Lys también se asocian a remodelación de la cromatina dependiente de ATP, alterando la posición de los nucleosomas y exponiendo genes a la transcripción.
La metilación es catalizada por metiltransferasa de histonas (MTH), correspondiendo a cambios epigenéticos. Pero también se ha puesto de manifiesto el fenómeno inverso de desmetilación de histonas, por el que la enzima amino oxidasa lisina desmetilasa específica 1 (LSD-1) y enzimas de la familia jumonji C (JmjC) (*) son capaces de desmetilar los resíduos Lys. Se trata de un proceso dinámico que puede alterar las tasas de transcripción génica a corto plazo.
(*) JmjC desmetilasas son oxigenasas dependientes de Fe (II) y α-cetoglutarato (αKG) para su actividad. La desmetilación de histonas a través de proteínas JmjC ocurre a través de una reacción de hidroxilación, en la que se utilizan αKG, oxígeno y Fe (II) para producir succinato y CO2. Esta reacción de hidroxilación da como resultado productos de hemiamina inestables, que se descomponen en formaldehído y un producto de histona desmetilada. Actualmente se sabe que veinticuatro enzimas humanas contienen un dominio JmjC y tienen actividad de histona desmetilasa. Estas enzimas se organizan en subfamilias basadas en la homología de secuencia y la preferencia del estado de metilación. Las histonas desmetilasas de JmjC regulan el estado de la cromatina mediante la eliminación de las marcas de mono, di y trimetilación en los residuos de lisina de múltiples histonas. Consistentemente, JmjC desmetilasas juegan un papel en diversas condiciones patológicas. Por ejemplo, JmjC desmetilasas han demostrado ser pro-tumorigénicas y supresoras de tumores. Los cambios dentro del estado de cromatina pueden conducir a la activación o represión de la transcripción, dependiendo del residuo de aminoácido al que se dirige. Además, los cambios de metilación también pueden afectar otros procesos celulares, como la reparación del daño del ADN, sin afectar la transcripción de los genes circundantes
La metilación de histonas puede ser dinámica o estable a lo largo de la vida de una célula e incluso a través de la mitosis y la meiosis y, en algunas ocasiones, puede heredarse de padres a hijos. Por otra parte, los niveles de metilación de histonas cambian con la edad, y se ha demostrado que las metiltransferasas y desmetilasas regulan la longevidad de varios organismos modelo. En algunos casos, se ha demostrado que estas enzimas tienen un efecto transgeneracional (52).
Otras modificaciones ejercen su efecto en gran medida al influir en los patrones de metilación o acetilación. Entre estas modificaciones están la ubiquitinación (*) de las histonas H2A, H2B y H3, que se correlaciona con una mayor transcripción génica.
(*) La ubiquitina (o ubicuitina) es una pequeña proteína reguladora que ha sido encontrada en la mayoría de los tejidos de los organismos eucariotas. Una de sus muchas funciones es dirigir el reciclaje de proteínas. La ubiquitina puede asociarse a proteínas y marcarlas para su destrucción. El marcaje de ubiquitina dirige las proteínas al proteosoma, que es un gran complejo de proteínas que encontramos en la célula y que degrada y recicla proteínas innecesarias.
La ubiquitinización es un proceso enzimático de modificación protéica post-traduccional en el cual el ácido carboxílico de la glicina terminal forma un enlace amida con el grupo amino épsilon de la lisina en la proteína modificada. La ubiquitinización funciona en una gran variedad de procesos celulares fisiológicos y patológicos.
Otros procesos como la fosforilación de histonas pueden evitar la acetilación de las mismas contribuyendo al mantenimiento de la conformación cerrada del ADN la heterocromatina. Las modificaciones covalentes de histonas (ubiquitinación y fosforilación) agregan un control dinámico adicional a los mecanismos ya versátiles de acetilación y metilación de histonas.
Las modificaciones de histonas intervienen en la determinación de la identidad de las células β. Diversos mecanismos pueden, al desactivar ciertos genes, aumentar la expresión de otros o predisponer genes de células no β para la metilación inhibitoria. Por ejemplo, dentro de la célula β, las histonas que regulan la expresión de la proteína homeobox arista-less (Arx) se metilan antes de la metilación del ADN para evitar un programa de diferenciación de células α. De hecho, existe una relación mutuamente antagónica entre Arx y Pax4, de modo que Pax4 debe expresarse y Arx suprimirse para especificar la identidad de las células (Figura 21).
La expresión y la actividad de HDAC también están reguladas en el desarrollo durante la organogénesis pancreática. Se ha demostrado que la administración de inhibidores de HDAC de molécula pequeña a explantes pancreáticos embrionarios suprime la diferenciación acinar, promueve la diferenciación ductal, aumenta la expresión de Pax4 y mejora el desarrollo del grupo de células β (Figuras 21 y 22).
En cuanto al gen regulador de la secreción de insulina, estudios realizados en células β de animales de experimentación, han mostrado la presencia de marcas de activación, en forma de hiperacetilación de H3 e hipermetilación de H3-K4, en el promotor proximal de insulina. En líneas celulares β, la cetilación de la histona H3 se relaciona con la acción de la histona acetil transferasa (HAT) p300 sobre el gen de insulina (Pdx-1). Un fenómeno ausente en relación con células α (53, 54, 56, 57).
Diversos estudios han puesto de manifiesto las consecuencias de la alteración de la cromatina en la función de las células β. Por ejemplo, la lipotoxicidad aguda y crónica inducida por palmitato, en una línea celular β clonal, provocó un aumento de la actividad de la HAT y cambios en la expresión de varios genes importantes de las células β. Ello se asoció a cambios coordinados en la activación o silenciamiento de los patrones de metilación y acetilación de histonas. En condiciones de hiperglucemia, se ha demostrado que los cambios en la acetilación de histonas activan vías beneficiosas y perjudiciales relacionadas con la supervivencia y función de las células β. Durante la hiperglucemia, la proteína p300 de HAT, a través de la acetilación de la histona H4 del gen TXNIP, conduce a una disminución de la actividad de tiorredoxina. En modelos de T1D y T2D, la disminución de la actividad de tiorredoxina aumentó las especies reactivas de oxígeno y la apoptosis de las células β (59)
- Diabetes y ncARN
Antes hemos señalado como los ARN no codificantes (ncARN) pueden regular la expresión génica al regular la síntesis proteica, a nivel postranscripcional y traduccional. Algunas variedades de ncARN podrían tener un papel importante en el desarrollo de las células β y en la biosíntesis y secreción de la insulina. Los miARN reducen la estabilidad o inhiben la traducción de transcripciones diana uniéndose a sus regiones 3'-no traducidas (UTR). Los lncRNA se expresan con especificidad de tejido y consisten en diferentes tipos de ARN funcionales que son capaces de reclutar DNMT y modificadores de histonas para sus genes objetivo. Existe evidencia de la regulación de la expresión génica específica de los islotes por los lncRNAs, aunque su papel en el desarrollo del páncreas y la función de las células β está por definir.
10. Dieta y epigenética
Ahora sabemos que tanto los seres humanos, como el resto de organismos vivos, no son solamente lo que está escrito en el genoma desde su concepción. Por el contrario, la investigación actual está muy interesada y dirigida a descubrir el código de pequeñas modificaciones químicas capaces de regular la expresión de muchos genes.
Se han descrito unos 100 loci con posible influencia en el desarrollo de la DM2. Sin embargo, sus variantes tienen un efecto reducido sobre el riesgo de DM2 y solo pueden explicar un 10% de la herencia de DM2. Ello es así porque las interacciones epigenéticas también influyen en la asociación estadística y real de un SNP con una enfermedad y también con la respuesta a la dieta.
Entramos directamente dentro de una disciplina en auge como es la Nutrigenómica (*). Pero profundizar en sus dominios nos llevaría muy lejos y nos saldríamos de los límites que este artículo, ya suficientemente profuso, puede incluir. Posiblemente es un tema que merecería una consideración a parte. Sin embargo, los conceptos generales nutrigenómicos pueden servirnos para aproximarnos a al trinomio: dieta, genética y diabetes.
(*) La genómica nutricional, o Nutrigenómica, es el estudio de cómo los alimentos afectan a la expresión de la información genética en los individuos y de cómo la composición genética de un individuo responde a nutrientes y sustancias bioactivas. No todos los individuos responden de la misma manera al alimento. Los individuos heredan una respuesta única a los alimentos y, dado que los alimentos influyen en la salud, una susceptibilidad única a las enfermedades crónicas. Las diferencias en susceptibilidad a los nutrientes son causadas por los mismos cambios genéticos que impulsan la evolución. La Nutrigenómica busca proporcionar una comprensión genética molecular de cómo los sustancias químicas dietéticas comunes (es decir, la nutrición) afectan la salud al alterar la expresión y / o la estructura de la composición genética de un individuo. La progresión de un fenotipo sano a un fenotipo de enfermedad crónica sería el resultado de una acción de los componentes químicos de la dieta, capaz de dar lugar a cambios en la expresión génica o a diferencias en las actividades de proteínas y enzimas.El concepto central de la Nutrigenómica es que el alimento altera la expresión de la información genética y que las diferencias genotípicas dan lugar a diferentes perfiles metabólicos. Y ello, de hecho, proporciona el vínculo crítico entre dieta y salud.
La base conceptual para esta nueva rama de la investigación genómica se centra en los principios de la genómica nutricional:
1) Las sustancias químicas presentes en los alimentos comunes actúan sobre el genoma humano, directa o indirectamente, para alterar la expresión o estructura génica.Estos principios se basan en datos procedentes de estudios genéticos, epidemiológicos, nutricionales, moleculares, fisiológicos y de otras fuentes científicas relacionadas con la investigación en salud (61).
2) Bajo ciertas circunstancias y en algunos individuos, la dieta puede ser un factor de riesgo grave para una serie de enfermedades.
3) Es probable que algunos genes regulados por la dieta (y sus variantes normales y comunes) desempeñen un papel en la aparición, incidencia, progresión y/o gravedad de enfermedades crónicas.
4) El grado en que la dieta influye en el equilibrio entre los estados de salud y enfermedad puede depender de la composición genética de un individuo.
5) la intervención dietética basada en el conocimiento de los requerimientos nutricionales, el estado nutricional y el genotipo (es decir, "nutrición individualizada") puede usarse para prevenir, mitigar o curar enfermedades crónicas.
No se puede descartar que la expresión de algunos de los genes implicados en la DM2 esté regulada por factores dietéticos. La susceptibilidad a padecer DM2 podría variar, dependiendo del tipo de alimentación, y ello podría modular la expresión de ciertos genes. Ejemplos de factores epigenéticos, ambientales y nutricionales, capaces de afectar al fenotipo de la DM2 son el contenido y el equilibrio energético de la ingesta, los patrones cronobiológicos, especialmente los referidos a los periodos sueño-vigilia, la actividad física, el nivel de hidratación y otros factores como el estrés (62).
Los nutrientes alteran los mecanismos epigenéticos a través de diferentes vías. En mamíferos (concretamente en ratones) se ha demostrado que una dieta rica en suplementos metilados aumentan la metilación de ADN y, lo que parece mas importante, cambian el fenotipo de la descendencia. El incremento del nivel de metilación se observó en los ratones sobre el locus del gen agouti (*) (63).
(*) El locus del gen agouti que controla la cantidad y distribución de pigmentación de eumelanina (marrón/negro) y feomelanina (amarillo/rojo) en el pelaje de los mamíferos. La mutación amarilla letal en el locus agouti fue la primera mutación letal embrionaria murina y el primer síndrome de obesidad murina que se caracterizó. El ratón agutí amarillo letal heterocigoto se caracteriza por un color de pelaje amarillo, obesidad de inicio tardío asociada con hiperfagia, hiperinsulinemia e hiperglucemia, aumento del crecimiento lineal y susceptibilidad a una amplia variedad de tumores epiteliales y mesenquimales. El gen agouti humano, que es 85% idéntico al gen del ratón, se expresa mucho más ampliamente, incluso en tejido adiposo, testículo, ovario y corazón, y en niveles más bajos en hígado, riñón y prepucio.Deficiencias dietéticas de colina, metionina, ácido fólico, vitamina B12, vitamina B6 y riboflavina alteran el metabolismo de un carbono (metabolismo de metilo) (*) y la concentración de uno de sus productos finales, S-adenosilmetionina (S-AM). S-AM es el precursor inmediato de las reacciones de metilación del ADN (Figura 23).
(*) El metabolismo de un carbono es una red de reacciones bioquímicas interrelacionadas que implican la transferencia de grupos de un carbono de un sitio a otro (Fig. 23). Hay un renovado interés en los componentes de la dieta que median, o de alguna manera facilitan, el metabolismo de un carbono. Los componentes dietéticos de interés son las vitaminas del grupo B, el folato y la vitamina B-12, así como la colina y la metionina. Colectivamente, estos nutrientes a veces se denominan “lipótropos dietéticos”, un término que surgió de la observación de que una dieta deficiente en colina sola, o colina en combinación con otros lipótropos, conduce a una acumulación excesiva de triglicéridos en el hígado de los roedores de laboratorio.La remodelación de la cromatina contribuye al control de la expresión génica. La estructura de la
La exposición a largo plazo a las dietas que influyen en la remodelación de la cromatina (p. ej., dietas hipercalóricas y la proteína de soja) y la metilación del ADN (metabolitos de un carbono) podrían inducir cambios epigenéticos permanentes en el genoma. Los cambios epigenéticos también pueden explicar la existencia de momentos clave durante el desarrollo. Es el caso de la vida intrauterina, en que las influencias ambientales a corto plazo pueden producir cambios duraderos en la expresión génica y el potencial metabólico.
11. Influencia epigenética de la vida intrauterina(*) En 1992, Hales y Barker acuñaron el término hipótesis del "fenotipo ahorrativo", derivado de la hipótesis previa del "genotipo ahorrativo". Neel propuso que los genes "ahorrativos" se seleccionaron durante la evolución en un momento en que los recursos alimentarios eran escasos y ello daba como resultado un "desencadenante rápido de insulina" y, por lo tanto, una mayor capacidad para almacenar grasa, lo que ponía al individuo en riesgo de resistencia a la insulina y a diabetes tipo 2 (67,68).
El concepto de que existen respuestas adaptativas embrionarias y fetales a un entorno intrauterino subóptimo que resultan en consecuencias adversas permanentes es consistente con la definición de "programación" de Lucas en 1991 como inducción, deleción o desarrollo deteriorado de un permanente estructura somática o el "ajuste" de un sistema fisiológico por un estímulo o insulto temprano que opera en un período "sensible", lo que resulta en consecuencias a largo plazo para la función (Figura 24). En el trabajo de Lucas se señala que, en la rata el período sensible para la programación nutricional del metabolismo del colesterol y el triacilglicerol es pre y postnatal (pre-destete) y que las ratas pueden programarse 'indirectamente' al alterarse el medio nutricional materno durante la gestación o la lactancia (69).
Se ha propuesto el término “plasticidad” en lugar de el de “programación” como más apropiado, para definir “la capacidad de un genotipo único para producir más de una forma alternativa de estructura, o dar lugar a un estado fisiológico o comportamiento, en respuesta a factores ambientales”. También se ha considerado que el concepto original de la hipótesis del fenotipo económico, que se aplica específicamente a las condiciones de privación intrauterina, puede ser demasiado limitado, como hipótesis, para explicar el desarrollo de la enfermedad en adultos y las respuestas adaptativas del embrión o el feto a una variedad de entornos prenatales. Por ello, debería considerarse un contexto evolutivo más amplio, para explicarlo (34,70).
Los cambios epigenéticos en respuesta a señales ambientales pueden comenzar incluso antes del nacimiento. El estado metabólico de los progenitores y el entorno intrauterino tienen una gran importancia en la conformación del epigenoma del sujeto adulto. La restricción del crecimiento intrauterino se produce por mala nutrición materna o por un déficit en intercambio de nutrientes a nivel de la placenta. En situación de IUGR los descendientes tienen mayor predisposición a la obesidad y a padecer DM2 en edad adulta. Se supone que los cambios epigenéticos desempeñan un papel destacado en estos trastornos metabólicos de susceptibilidad observados.
Mas arriba nos hemos referido a la influencia de la IUGR, a través de la acetilación de histonas. En ratones, se ha observado un mecanismo epigenético, mediado por una alteración en la acetilación de histonas de nucleosomas relacionados con el gen Pdx1, que lleva a la represión de la expresión de dicho gen (Figura 19).
Los efectos específicos sobre las células β, en situación de IUGR se han estudiado utilizando modelos en roedores, mediante ligadura bilateral de arterias uterinas. Ello reduce el flujo sanguíneo placentario en cerca de un 50%. Como consecuencia se produjo una descendencia con bajo peso al nacer. A las 7 semanas, la descendencia de las madres tratadas con ligadura bilateral de arterias uterinas presentaban hiperglucemia leve, hiperinsulinemia y reducción de la masa de células β. A las 26 semanas de edad, los cachorros de IUGR presentaban obesidad franca e hiperglucemia severa, con alteraciones aún más significativas en la masa de células β. Además, los islotes de descendientes de IUGR tenían una expresión reducida de Pdx-1, asociada a la desacetilación de H3 y H4, la desmetilación de H3K4 y el aumento de la metilación de H3K9 en el promotor Pdx-1 (46, 71, 72)
La persistencia de la disminución de la función pancreática en la vida postnatal, cuando el entorno nutricional postnatal se corresponde con el entorno nutricional prenatal, sugiere que la mala nutrición materna tiene un efecto adverso sobre la capacidad funcional pancreática como consecuencia de la aparición de un déficit irreversible del desarrollo (Figura 25).
Se ha propuesto el término “plasticidad” en lugar de el de “programación” como más apropiado, para definir “la capacidad de un genotipo único para producir más de una forma alternativa de estructura, o dar lugar a un estado fisiológico o comportamiento, en respuesta a factores ambientales”. También se ha considerado que el concepto original de la hipótesis del fenotipo económico, que se aplica específicamente a las condiciones de privación intrauterina, puede ser demasiado limitado, como hipótesis, para explicar el desarrollo de la enfermedad en adultos y las respuestas adaptativas del embrión o el feto a una variedad de entornos prenatales. Por ello, debería considerarse un contexto evolutivo más amplio, para explicarlo (34,70).
Los cambios epigenéticos en respuesta a señales ambientales pueden comenzar incluso antes del nacimiento. El estado metabólico de los progenitores y el entorno intrauterino tienen una gran importancia en la conformación del epigenoma del sujeto adulto. La restricción del crecimiento intrauterino se produce por mala nutrición materna o por un déficit en intercambio de nutrientes a nivel de la placenta. En situación de IUGR los descendientes tienen mayor predisposición a la obesidad y a padecer DM2 en edad adulta. Se supone que los cambios epigenéticos desempeñan un papel destacado en estos trastornos metabólicos de susceptibilidad observados.
Mas arriba nos hemos referido a la influencia de la IUGR, a través de la acetilación de histonas. En ratones, se ha observado un mecanismo epigenético, mediado por una alteración en la acetilación de histonas de nucleosomas relacionados con el gen Pdx1, que lleva a la represión de la expresión de dicho gen (Figura 19).
Los efectos específicos sobre las células β, en situación de IUGR se han estudiado utilizando modelos en roedores, mediante ligadura bilateral de arterias uterinas. Ello reduce el flujo sanguíneo placentario en cerca de un 50%. Como consecuencia se produjo una descendencia con bajo peso al nacer. A las 7 semanas, la descendencia de las madres tratadas con ligadura bilateral de arterias uterinas presentaban hiperglucemia leve, hiperinsulinemia y reducción de la masa de células β. A las 26 semanas de edad, los cachorros de IUGR presentaban obesidad franca e hiperglucemia severa, con alteraciones aún más significativas en la masa de células β. Además, los islotes de descendientes de IUGR tenían una expresión reducida de Pdx-1, asociada a la desacetilación de H3 y H4, la desmetilación de H3K4 y el aumento de la metilación de H3K9 en el promotor Pdx-1 (46, 71, 72)
La persistencia de la disminución de la función pancreática en la vida postnatal, cuando el entorno nutricional postnatal se corresponde con el entorno nutricional prenatal, sugiere que la mala nutrición materna tiene un efecto adverso sobre la capacidad funcional pancreática como consecuencia de la aparición de un déficit irreversible del desarrollo (Figura 25).
(*) La vía de señalización JAK/STAT involucra tres elementos: 1. Receptor de ligando, la mayoría son receptores de citoquinas, de ahí la función de esta vía en la respuesta inmunológica. 2. Proteínas JAK asociadas al receptor en el interior de la célula y 3.-Proteínas STAT (Signal Transducer and Activator of Transcription), cuya función es la de actuar como factores de transcripción una vez activadas por fosforilación.
 |
| Figura 26: Vía de señalización JAK/STAT. Autor: Peter Znamenkiy (2006). Wikipedia. Public domain |
12. Herencia epigenética transgeneracional en DM2
 |
| Figura 27: Efectos epigenéticos transgeneracionales e intergeneracionales. Heard E, Martienssen RA. Transgenerational epigenetic inheritance: myths and mechanisms. Cell 2014; 157:95-109 |
En mamíferos, los cambios epigenéticos pueden ser inducidos por el medio ambiente (toxinas, nutrición, estrés). En roedores, en el caso de una hembra preñada expuesta a una influencia ambiental, el feto puede verse afectado en el útero (F1), al igual que la línea germinal del feto (el futuro F2). Son efectos parentales, con herencia epigenética intergeneracional (figura 27). Como se expone en la figura, solamente los individuos F3 podrían considerarse para una herencia transgeneracional verdadera, en ausencia de exposición a factores ambientales. El caso del varón es diferente: ante un cambio epigenético, el individuo (F0) y su línea germinal (futura F1) están expuestos. El F1 se considera así como intergeneracional, mientras que solo F2 y generaciones posteriores podrían considerarse como herencia transgeneracional.
Herencia epigenética y reprogramación de la línea germinal son dos mecanismos complementarios y encontrados. La reprogramación de la línea germinal es un fenómeno importante en la biología del desarrollo. Es un mecanismo que permite eliminar firmas epigenéticas adquiridas durante el desarrollo o relacionadas con el medio ambiente. El objetivo es que el embrión refleje adecuadamente el patrón genético característico de cada especie. Si no funcionara el mecanismo de reprogramación, las marcas epigenéticas pueden mantenerse y transmitirse de una generación a la siguiente.
En ratones hay al menos dos ventanas de reprogramación de metilación de ADN en todo el genoma. La primera tiene lugar después de la fertilización, en las etapas de cigoto y división temprana del mismo, para borrar las marcas epigenómicas de los gametos (espermatozoides y ovocitos). Sin embargo, durante esta fase, se mantienen las huellas genómicas. La otra ventana importante de reprogramación ocurre en la línea germinal, para borrar programas somáticos paternos y maternos, además de las huellas genómicas y tiene lugar la reactivación del segundo cromosoma X (*). Mas tarde, las huellas específicas de los padres se establecen en la línea germinal. En cada ventana de reprogramación, un conjunto específico de mecanismos regula el borrado y el restablecimiento de la metilación del ADN (Figura 27) (75).
 |
| Figura 28: Reprogramación de la línea germinal de la metilación del ADN en ratones. Heard E, Martienssen RA. Transgenerational epigenetic inheritance: myths and mechanisms. Cell 2014; 157:95-109 |
(*) Para compensar la carga genómica de los genes localizados en el cromosoma X entre ambos sexos biológicos (las mujeres tienen dos cromosomas X y los hombres un único cromosoma X) cada célula femenina inactiva aleatoriamente uno de sus cromosomas X. El proceso de inactivación se inicia durante el desarrollo temprano, momento en el que las células toman la decisión de qué cromosoma X se inactiva, si el paterno o el materno. Posteriormente, esta decisión se mantiene mediante mecanismos epigenéticos en las células hijas y a través de las sucesivas divisiones.Como ocurre con las mutaciones de la secuencia de ADN, la mayoría de las "mutaciones" epigenéticas, también denominadas “epialelos”, pueden no tener repercusión funcional o tener efectos deletéreos en la expresión genética. Además, estos epialelos pueden dar lugar a la liberación de elementos transponibles y a la liberación de elementos que se han denominado “parásitos genómicos”.
Por ejemplo, se ha observado que la presencia de copias adicionales del cromosoma X está asociada a un mayor riesgo a desarrollar lupus eritematoso en aquellas personas portadoras, mientras que en las personas con síndrome de Turner (que tienen un único cromosoma X) hay un menor riesgo a de padecerla. En este sentido, se ha estudiado la inactivación del cromosoma X en los linfocitos T (con un papel relevante en enfermedades autoinmunes como el lupus), tanto en ratones y como en células de pacientes con lupus. Los investigadores observaron la localización del ARN Xist (ncARN con un papel esencial en la inactivación del cromosoma X), que tras ser expresado por el cromosoma que será inactivado, recluta diferentes proteínas hacia este cromosoma, que intervienen en su silenciamiento. Durante el desarrollo el ARN Xist se localiza más alejado del cromosoma X, mientras que cuando los linfocitos T son activados, este ARN se localiza concentrado en el cromosoma X inactivado. Está en estudio si la alteración de los complejos proteicos, que interaccionan con ARN Xist, aumentan la predisposición al lupus u otras enfermedades autoinmunes (76).
En cualquier caso, la herencia epigenética transgeneracional también tiene el potencial de ser adaptativa e incluso podría responder a desafíos ambientales, con importantes implicaciones para la herencia, la reproducción y la evolución.
Se han documentado ejemplos potenciales de herencia epigenética; sin embargo, los estudios que consideran esta posibilidad suelen referirse a efectos intergeneracionales en lugar de transgeneracionales, pero no excluyen cambios genómicos en la secuencia de ADN como causa de la transcendencia hereditaria.
En mamíferos existen dos fases importantes de reprogramación: en la fase embrionaria y en la línea germinal. Estos mecanismos de “borrado” de marcas epigenéticas dejan pocas posibilidades para la herencia de marcas epigenéticas, sean del tipo que sean (programadas, accidentales o inducidas por factores ambientales). Es cierto que la herencia de un “estado adquirido” puede darse en invertebrados (nematodos), pero en mamíferos está por demostrar (77).
Sin embargo, existe una creciente evidencia, en diferentes modelos, que demuestran que la herencia transgeneracional de riesgo de enfermedad puede tener efectos fenotípicos paralelos a los factores de riesgo heredados de manera clásica o mendeliana. Ello, además de una evidencia sugestiva de herencia transgeneracional en humanos, sugiere que este modo de herencia puede contribuir más, de lo hasta ahora aceptado, a la susceptibilidad a las enfermedades metabólicas (74).
Por ejemplo, la exposición de ratones preñados a una dieta baja en proteínas perjudica el desarrollo del páncreas endocrino en sus crías. Y hay evidencia de que este fenómeno puede persistir en generaciones posteriores. Algunos estudios han observado que la restricción de proteínas en ratones durante el embarazo produce cambios morfológicos en los islotes pancreáticos, lo que sugiere que la homeostasis de la glucosa se mantiene por una mayor sensibilidad a la insulina durante las primeras etapas de la vida en la descendencia durante tres generaciones consecutivas (78).Existen otros estudios que indican que la prediabetes paterna aumenta la susceptibilidad a la diabetes en la descendencia a través de alteraciones epigenéticas de los gametos. En estas investigaciones, la prediabetes paterna condujo a intolerancia a la glucosa y resistencia a la insulina en la descendencia. En relación con los controles, la descendencia de los padres prediabéticos mostraba patrones de expresión génica alterados en islotes pancreáticos, con baja regulación de varios genes implicados en el metabolismo de la glucosa y las vías de señalización de la insulina. El perfil epigenómico de los islotes pancreáticos de descendientes reveló numerosos cambios en la metilación de citosina dependientes de la prediabetes paterna, incluidos cambios reproducibles en la metilación en varios genes de señalización de insulina. Estos estudios parecen revelar que la prediabetes se puede heredar transgeneracionalmente a través de la línea germinal de los mamíferos, mediante un mecanismo epigenético, incluyendo cambios reproducibles en la metilación sobre varios genes de señalización de insulina (79).
Un interesante estudio epidemiológico realizado sobre población sueca en 2002 se plantea cuestiones como que la disponibilidad de alimentos podría conducir a la sobrealimentación y, a su vez, a adaptaciones metabólicas. Pero lo mas interesante de su planteamiento es la pregunta: “Además de la selección, ¿podrían estas adaptaciones al entorno social tener efectos transgeneracionales?“. Concretamente, los investigadores, se cuestionan si comer en exceso, durante el período de crecimiento lento (PCL) de un niño, antes del pico prepuberal, puede influir en el riesgo de muerte de los descendientes por enfermedad cardiovascular y diabetes.
En este estudio (cuyos datos se recopilaron siguiendo a tres cohortes nacidas en 1890, 1905 y 1920 en la parroquia de Överkalix en el norte de Suecia hasta la muerte o hasta el año 1995), el acceso de padres o abuelos a los alimentos durante su PCL se determinó mediante datos históricos sobre cosechas y precios de los mismos, registros de la comunidad local y hechos históricos generales. Si la comida no estaba fácilmente disponible durante el PCL del padre, la mortalidad por enfermedad cardiovascular del de los descendientes era baja. La mortalidad por diabetes aumentó si el abuelo paterno estuvo expuesto a un exceso de alimentos durante su PCL (Odds Ratio 4.1, IC del 95% 1.33–12.93, P= 0,01). Por tanto, un mecanismo relacionado con la nutrición, a través de la línea masculina parece haber influido en el riesgo de mortalidad cardiovascular y diabetes mellitus.El resultado más llamativo es que, las circunstancias relacionadas con la nutrición del entorno social, tenían repercusiones transgeneracionales, con muertes cardiovasculares y relacionadas con la diabetes y que, estas repercusiones en la herencia, se circunscribieron a la línea masculina:
Cuando el padre (P = 0.05) y tal vez la abuela paterna (P = 0.11) estuvieron expuestos a una hambruna durante su PCL, el descendiente estaba protegido contra las causas cardiovasculares de muerte. Además, si el abuelo paterno vivió una hambruna durante su PCL, tendió a proteger al paciente de la diabetes (P = 0.09). Sin embargo, lo más interesante fue el hallazgo de que si los abuelos paternos tenían acceso a un exceso de alimentos durante su PEC, sus nietos tenían un riesgo cuatro veces mayor de muerte por diabetes mellitus según la estimación puntual (P = 0.01).
Los autores especulan sobre el origen del aumento de riesgo en relación con mortalidad y diabetes, atribuyéndola a la variación en el minisatélite de repetición en tándem de número variable (VNTR) 5' (*) del gen de insulina que está asociado con la DM1. La variación, más plausible en el VNTR, influye también en la susceptibilidad a la diabetes tipo 2.
En este estudio (cuyos datos se recopilaron siguiendo a tres cohortes nacidas en 1890, 1905 y 1920 en la parroquia de Överkalix en el norte de Suecia hasta la muerte o hasta el año 1995), el acceso de padres o abuelos a los alimentos durante su PCL se determinó mediante datos históricos sobre cosechas y precios de los mismos, registros de la comunidad local y hechos históricos generales. Si la comida no estaba fácilmente disponible durante el PCL del padre, la mortalidad por enfermedad cardiovascular del de los descendientes era baja. La mortalidad por diabetes aumentó si el abuelo paterno estuvo expuesto a un exceso de alimentos durante su PCL (Odds Ratio 4.1, IC del 95% 1.33–12.93, P= 0,01). Por tanto, un mecanismo relacionado con la nutrición, a través de la línea masculina parece haber influido en el riesgo de mortalidad cardiovascular y diabetes mellitus.El resultado más llamativo es que, las circunstancias relacionadas con la nutrición del entorno social, tenían repercusiones transgeneracionales, con muertes cardiovasculares y relacionadas con la diabetes y que, estas repercusiones en la herencia, se circunscribieron a la línea masculina:
Cuando el padre (P = 0.05) y tal vez la abuela paterna (P = 0.11) estuvieron expuestos a una hambruna durante su PCL, el descendiente estaba protegido contra las causas cardiovasculares de muerte. Además, si el abuelo paterno vivió una hambruna durante su PCL, tendió a proteger al paciente de la diabetes (P = 0.09). Sin embargo, lo más interesante fue el hallazgo de que si los abuelos paternos tenían acceso a un exceso de alimentos durante su PEC, sus nietos tenían un riesgo cuatro veces mayor de muerte por diabetes mellitus según la estimación puntual (P = 0.01).
Los autores especulan sobre el origen del aumento de riesgo en relación con mortalidad y diabetes, atribuyéndola a la variación en el minisatélite de repetición en tándem de número variable (VNTR) 5' (*) del gen de insulina que está asociado con la DM1. La variación, más plausible en el VNTR, influye también en la susceptibilidad a la diabetes tipo 2.
(*) En Genética molecular, el número variable de repeticiones en tándem (del inglés Variable Number of Tandem Repeats y más conocidas por el acrónimo VNTR) o minisatélites, son repeticiones de secuencias de 9 a 100 pares de bases que se utilizan como marcador molecular. El número de repeticiones es variable pero en general es menor a 1000.Este efecto está mediado exclusivamente por el alelo derivado de la madre, que implica un gen impreso (posiblemente IGF2) en la patogénesis de la diabetes tipo 2.22 Ins2 en ratones e IGF2 (Ver mas arriba en Apartado 5. Genes de riesgo para la diabetes: IGF2BP2, proteína 2 de unión al ARNm del factor de crecimiento similar a la insulina 2, que codifica una proteína que se une a la 5'-UTR) en el saco vitelino también se expresan únicamente a partir del alelo paterno que insinúa más tarde la insulina y las enfermedades relacionadas con el crecimiento.
Se plantea la cuestión de si el efecto transgeneracional relacionado con la nutrición en la línea masculina implica la modificación epigenética directa del ADN y/o las histonas asociadas. Se ha argumentado que los genes impresos (*) son buenos candidatos para mediar tales efectos transgeneracionales.
(*) La impronta genómica puede definirse como la expresión selectiva de un gen según el origen parental del alelo (paterno o materno). En los organismos diploides (como los humanos), las células somáticas poseen dos copias del genoma, una heredada del padre y otra de la madre. Por lo tanto, cada gen autosómico está representado por dos copias, o alelos, con una copia heredada de cada padre en la fertilización. Para la gran mayoría de los genes autosómicos, la expresión se produce en ambos alelos simultáneamente. Sin embargo, en los mamíferos, una pequeña proporción (<1%) de los genes están impresos, lo que significa que la expresión génica se produce a partir de un solo alelo. El alelo expresado depende de su origen parental. Por ejemplo, el gen que codifica el factor de crecimiento similar a la insulina 2 (IGF2) solo se expresa a partir del alelo heredado del padre.En este sentido se ha propuesto que la modulación transgeneracional de la expresión génica sería posible, si la respuesta metabólica de los padres a algún estrés fisiológico o social modificara la situación de impronta. Los reguladores de la transcripción podrían teóricamente mediar en este proceso. La naturaleza funcional de los genes impresos, entre un estado transcripcionalmente activo y silencioso, los hace buenos candidatos para su incorporación a la evolución de los sistemas de adaptación transgeneracional, donde los cambios coordinados en la expresión génica a lo largo de las generaciones es una ventaja selectiva (80,81).
REFERENCIAS:
- N. Carrera Cachaza. Estudio genético en esquizofrenia: análisis de variantes funcionales para la identificación de factores de predisposición. Memoria para optar al grado de Doctor. Universidade de Santiago de Compostela. Facultade de Medicina. Departamento de Anatomía Patoloxica e Ciencias Forenses. Enero 2012.
- Tuuli Lappalainen, Michael Sammeth, Marc R Friedländer, et al.. Transcriptome and genome sequencing uncovers functional variation in humans. Nature. 2013 Sep 26; 501(7468): 506–511.
- Masters SL, et al. Activation of the NLRP3 inflammasome by islet amyloid polypeptide provides a mechanism for enhanced IL-1beta in type 2 diabetes. Nat Immunol. 2010;11:897–904.
- Wolford JK1, Vozarova de Courten B. Genetic basis of type 2 diabetes mellitus: implications for therapy. Treat Endocrinol. 2004; 3(4):257-67
- R. Barrio. Diabetes monogénicas: enfoque diagnóstico y tipos más frecuentes. Av Diabetol. 2007; 23(5):333-340
- Eric S. Lander. The New Genomics: Global Views of Biology. Science 1996; 274,(5287):536-539.
- Vaxillaire M, Bonnefond A, Froguel P. The lessons of early-onset monogenic diabetes for the understanding of diabetes pathogenesis. Best Pract Res Clin Endocrinol Metab. 2012; 26(2):171-87.
- Florez JC, Hirschhorn J, Altshuler D. The inherited basis of diabetes mellitus: implications for the genetic analysis of complex traits. Annu Rev Genomics Hum Genet. 2003; 4: 257-91.
- Voight BF, Scott LJ, Steinthorsdottir V, et al.. Twelve type 2 diabetes susceptibility loci identified through large-scale association analysis. Nat Genet. 2010; 42(7):579-89.
- Sladek R1, Rocheleau G, Rung J, et al.. A genome-wide association study identifies novel risk loci for type 2 diabetes. Nature. 2007; 445(7130):881-5.
- Wellcome Trust Case Control Consortium. Genome-wide association study of 14,000 cases of seven common diseases and 3,000 shared controls. Nature. 2007;447(7145):661-78.
- Peng S1, Zhu Y, Lü B, Xu F, Li X, Lai M. TCF7L2 gene polymorphisms and type 2 diabetes risk: a comprehensive and updated meta-analysis involving 121,174 subjects. Mutagenesis. 2013 Jan;28(1):25-37.
- Lyssenko V, Lupi R, Marchetti P, Del Guerra S, et al.. Mechanisms by which common variants in the TCF7L2 gene increase risk of type 2 diabetes. J Clin Invest. 2007; 117(8):2155-63.
- Florez JC, Jablonski KA, Bayley N, et al. TCF7L2 polymorphisms and progression to diabetes in the Diabetes Prevention Program. N Engl J Med 2006;355:241–250.
- Vcelak J, Vejrakova D, Vaňkova M, Lukasova P, Bradnova O, Halkova T, Bestak J, Andělova K, Kvasnickova H, Hoskovcova P, et al. T2d risk haplotypes of the tcf7l2 gene in the czech population sample: the association with free fatty acids composition. Physiol Psychol. 2012; 61(3):229.
- Grant SF, Thorleifsson G, Reynisdottir I, Benediktsson R, Manolescu A, Sainz J, Helgason A, Stefansson H, Emilsson V, Helgadottir A, et al. Variant of transcription factor 7-like 2 (tcf7l2) gene confers risk of type 2 diabetes. Nat Genet. 2006;38(3):320–3.
- Cauchi S, Meyre D, Dina C, et al.. Transcription factor TCF7L2 genetic study in the French population: expression in human beta-cells and adipose tissue and strong association with type 2 diabetes. Diabetes, 2006;55(10):2903-8.
- Osmark P, Hansson O, Jonsson AM, Ronn T, Groop L, Renstrom E. Unique splicing pattern of the tcf7l2 gene in human pancreatic islets. Diabetologia. 2009;52(5):850–4.
- Weiyue Ding, Li Xu, Lejun Zhang, Zhijie Han, Qinghua Jiang, Zhe Wang, y Shuilin Jin. Meta-analysis of association between TCF7L2 polymorphism rs7903146 and type 2 diabetes mellitus. BMC Med Genet, 2018; 19:38-50.
- Kaput J, Dawson K. Complexity of Type 2 Diabetes Mellitus Data Sets Emerging from Nutrigenomic Research: A Case for Dimensionality Reduction? Mutat Res. 2007; 622(1-2): 19–32. http://www.ncbi.nlm.nih.gov/pmc/articles/PMC1994901/
- Ali O. Genetics of type 2 diabetes. World J Diabetes. 2013; 4(4):114-23.
- Struan F A Grant, Gudmar Thorleifsson, Kari Stefansson. Variant of transcription factor 7-like 2 (TCF7L2) gene confers risk of type 2 diabetes. Nature Genetics 2006; 38:320–323.
- Yang WS, Hsiung CA, Ho LT, et al.. Genetic epistasis of adiponectin and PPARgamma2 genotypes in modulation of insulin sensitivity: a family-based association study. Diabetologia. 2003; 46(7):977-83.
- Christensen BC1, Houseman EA, Marsit CJ, et al.. Aging and environmental exposures alter tissue-specific DNA methylation dependent upon CpG island context. PLoS Genet. 2009; 5(8):e1000602.
- Deaton AM, Bird A.. CpG islands and the regulation of transcription. Genes Dev. 2011; 25(10):1010-22.
- Steve Horvath. DNA methylation age of human tissues and cell types. Genome Biol. 2013; 14(10): R115.
- Sommese L, Zullo A, Mancini FP, Fabbricini R, Soricelli A, Napoli C. Clinical relevance of epigenetics in the onset and management of type 2 diabetes mellitus. Epigenetics. 2017 Jun 3; 12(6):401-415.
- Han P, Chang CP. Long non-coding RNA and chromatin remodeling. RNA Biol. 2015; 12(10):1094-8.
- Han P, Li W, Lin CH, et al.. A long noncoding RNA protects the heart from pathological hypertrophy. Nature. 2014; 514(7520):102-106.
- Zhu Y, Rowley MJ, Böhmdorfer G, Wierzbicki AT. A SWI/SNF chromatin-remodeling complex acts in noncoding RNA-mediated transcriptional silencing. Mol Cell. 2013; 49(2):298-309.
- Esteller M. Non-coding RNAs in human disease. Nat Rev Genet. 2011; 12(12):861-74.
- Mocellin, S., Provenzano, M. RNA interference: learning gene knock-down from cell physiology. J Transl Med 2004; 2:39. doi:10.1186/1479-5876-2-39.
- Hangauer MJ1, Vaughn IW, McManus MT. Pervasive transcription of the human genome produces thousands of previously unidentified long intergenic noncoding RNAs. PLoS Genet. 2013; 9(6):e1003569.
- McMillen IC, Robinson JS. Orígenes del desarrollo del síndrome metabólico: predicción, plasticidad y programación. Physiol.Rev. 2005; 85 (2): 571-633.
- Zoghbi HY1, Beaudet AL. Epigenetics and Human Disease. Cold Spring Harb Perspect Biol. 2016; 8(2):a019497.
- Sathishkumar C, Prabu P, Mohan V, Balasubramanyam M. Linking a role of lncRNAs (long non-coding RNAs) with insulin resistance, accelerated senescence, and inflammation in patients with type 2 diabetes. Hum Genomics. 2018; 12(1):41-49.
- Temelkova-Kurktschiev T, Stefanov T. Lifestyle and genetics in obesity and type 2 diabetes. Exp Clin Endocrinol Diabetes 2012; 120(01):1-6
- Sommese L, Zullo A, Mancini FP, Fabbricini R, Soricelli A, Napoli C. Clinical relevance of epigenetics in the onset and management of type 2 diabetes mellitus. Epigenetics. 2017 Jun 3; 12(6):401-415.
- Johnson JS, Evans-Molina C. Translational implications of the β-cell epigenome in diabetes mellitus. Transl Res. 2015; 165(1):91-101.
- Volkmar M, Dedeurwaerder S, Cunha DA, et al.. DNA methylation profiling identifies epigenetic dysregulation in pancreatic islets from type 2 diabetic patients. EMBO J. 2012; 31(6):1405-26.
- Rojas, A., Khoo, A., Tejedo, J. R., Bedoya, F. J., Soria, B., Martin, F.. Islet cell development. Adv. Exp. Med. Biol. 2010; 654: 59-75.
- Al-Hasani K, Pfeifer A, Courtney M, et al. Adult duct-lining cells can reprogram into beta-like cells able to counter repeated cycles of toxin-induced diabetes. Developmental cell. 2013; 26:86–100.
- Brun T, Franklin I, St-Onge L, et al. The diabetes-linked transcription factor PAX4 promotes {beta}-cell proliferation and survival in rat and human islets. J Cell Biol. 2004; 167(6):1123–1135.
- Talchai C1, Xuan S, Lin HV, Sussel L, Accili D. Pancreatic β cell dedifferentiation as a mechanism of diabetic β cell failure. Cell. 2012; 150(6):1223-34.
- Turner BM. Cellular memory and the histone code. Cell. 2002; 111(3):285-91.
- Park JH, Stoffers DA, Nicholls RD, Simmons RA. Development of type 2 diabetes following intrauterine growth retardation in rats is associated with progressive epigenetic silencing of Pdx1. J Clin Invest. 2008;118:2316–24.
- Lundh M, Christensen DP, Rasmussen DN, et al. Lysine deacetylases are produced in pancreatic beta cells and are differentially regulated by proinflammatory cytokines. Diabetologia. 2010; 53: 2569-2578.
- Christensen DP, Dahllof M, Lundh M, et al. Histone deacetylase (HDAC) inhibition as a novel treatment for diabetes mellitus. Mol Med. 2011;17:378–390.
- Miao F, Gonzalo IG, Lanting L, Natarajan R. In vivo chromatin remodeling events leading to inflammatory gene transcription under diabetic conditions. J Biol Chem. 2004;279:18091–7
- Lee HB, Noh H, Seo JY, Yu MR, Ha H. Histone deacetylase inhibitors: a novel class of therapeutic agents in diabetic nephropathy. Kidney Int Suppl. 2007:S61–6
- Villeneuve LM, Natarajan R. The role of epigenetics in the pathology of diabetic complications. Am J Physiol Renal Physiol 2010; 299(1):F14-25.
- Greer EL, Shi Y. Histone methylation: a dynamic mark in health, disease and inheritance. Nat Rev Genet. 2012;13(5):343-57.
- Chakrabarti SK, Francis J, Ziesmann SM, Garmey JC, Mirmira RG. Covalent histone modifications underlie the developmental regulation of insulin gene transcription in pancreatic beta cells. J Biol Chem. 27 de junio de 2003; 278 (26): 23617-23.
- Greer EL, Shi Y. Histone methylation: a dynamic mark in health, disease and inheritance. Nat Rev Genet. 2012;13(5):343-57.
- Dhawan S, Georgia S, Tschen SI, Fan G, Bhushan A. Pancreatic β cell identity is maintained by DNA methylation-mediated repression of Arx. Dev Cell. 2011; 20(4):419-29.
- Collombat P, Hecksher-Sørensen J, Krull J, Berger J, Riedel D, Herrera PL, Serup P, Mansouri A. Embryonic endocrine pancreas and mature beta cells acquire alpha and PP cell phenotypes upon Arx misexpression. J Clin Invest. 2007; 117(4):961-70.
- Haumaitre C, Lenoir O, Scharfmann R. Histone deacetylase inhibitors modify pancreatic cell fate determination and amplify endocrine progenitors. Mol Cell Biol. 2008; 28(20):6373-83.
- Bhandare R, Schug J, Le Lay J, Fox A, Smirnova O, Liu C, Naji A, Kaestner KH. Genome-wide analysis of histone modifications in human pancreatic islets. Genome Res. 2010; 20(4):428-33.
- Malmgren S, Spégel P, Danielsson AP, Nagorny CL, Andersson LE, Nitert MD, Ridderstråle M, Mulder H, Ling C. Coordinate changes in histone modifications, mRNA levels, and metabolite profiles in clonal INS-1 832/13 β-cells accompany functional adaptations to lipotoxicity. J Biol Chem. 2013; 288(17):11973-87.
- Arnes L, Sussel L. Epigenetic modifications and long noncoding RNAs influence pancreas development and function. Trends Genet. 2015; 31(6):290-9.
- Kaput J, Rodriguez RL. Nutritional genomics: the next frontier in the postgenomic era. Physiol Genomics. 2004;16(2):166-77.
- Stančáková A, Laakso M. Genetics of Type 2 Diabetes. Endocr Dev 2016; 31:203-20.
- Cooney CA, Dave AA, Wolff GL. Maternal methyl supplements in mice affect epigenetic variation and DNA methylation of offspring. J Nutr. 2002; 132(8 Suppl):2393S-2400S.
- Mason JB. Biomarkers of nutrient exposure and status in one-carbon (methyl) metabolism. J Nutr. 2003; 133 Suppl 3(3):941S-947S.
- Blander G, Guarente L. The Sir2 family of protein deacetylases. Annu Rev Biochem. 2004; 73: 417-35.
- Magbanua MJM, Dawson K, Huang L, Malyj W, Gregg J, Galvez A, Rodriguez RL. Nutrient - Gene Interactions involving soy Peptide and Chemopreventive Genes in Prostate Epithelial Cells. In: Kaput J, Rodriguez RL, editors. Nutritional Genomics Discovering the Path to Personalized Nutrition. John Wiley and Sons; Hoboken, NJ: 2006. pp. 255–276.
- Hales CN, Barker DJ. Type 2 (non-insulin-dependent) diabetes mellitus: the thrifty phenotype hypothesis. Diabetologia.1992; 35(7):595-601.
- Neel JV. Diabetes mellitus: a "thrifty" genotype rendered detrimental by "progress"? Am J Hum Genet. 1962; 14:353-62.
- Lucas A, Baker BA, Desai M, Hales CN. Nutrition in pregnant or lactating rats programs lipid metabolism in the offspring. Br J Nutr. 1996; 76(4):605-12.
- Barker DJ. Developmental origins of adult health and disease. J Epidemiol Community Health. 2004; 58(2):114-5.
- Simmons RA, Templeton LJ, Gertz SJ. Intrauterine growth retardation leads to the development of type 2 diabetes in the rat. Diabetes. 2001; 50(10):2279-86.
- Thompson RF, Fazzari MJ, Niu H, Barzilai N, Simmons RA, Greally JM. Experimental intrauterine growth restriction induces alterations in DNA methylation and gene expression in pancreatic islets of rats. J Biol Chem. 2010 ; 285(20):15111-8.
- Ng SF, Lin RC, Laybutt DR, Barres R, Owens JA, Morris MJ. Chronic high-fat diet in fathers programs β-cell dysfunction in female rat offspring. Nature. 2010; 467(7318):963-6.
- Stegemann R, Buchner DA. Transgenerational inheritance of metabolic disease. Semin Cell Dev Biol. 2015; 43:131-140.
- Seisenberger S, Peat JR, Hore TA, Santos F, Dean W, Reik W. Reprogramming DNA methylation in the mammalian life cycle: building and breaking epigenetic barriers. Philos Trans R Soc Lond B Biol Sci. 2013; 368(1609):20110330.
- Syrett CM, et al. Altered X-chromosome inactivation in T cells may promote sex-biased autoimmune diseases. JCI Insight. 2019.
- Heard E, Martienssen RA. Transgenerational epigenetic inheritance: myths and mechanisms. Cell 2014; 157:95-109.
- Frantz ED, Aguila MB, Pinheiro-Mulder Ada R, Mandarim-de-Lacerda CA. Transgenerational endocrine pancreatic adaptation in mice from maternal protein restriction in utero. Mech Ageing Dev. 2011; 132(3):110-6.
- Wei Y, Yang C-R, Wei YP, Zhao ZA, Hou Y, Schatten H, Sun QY. Herencia epigenética transgeneracional de DM2. Proc Natl Acad Sci U S A. 2014; 111(5):1873–1878.
- Pembrey M. Imprinting and transgenerational modulation of gene expression; human growth as a model. Acta geneticae medicae et gemellologiae, 1996; 45(1-2):111-25.
- Kaati G, Bygren LO, Edvinsson S. Cardiovascular and diabetes mortality determined by nutrition during parents' and grandparents' slow growth period. Eur J Hum Genet. 2002; 10(11):682-8.



















No hay comentarios:
Publicar un comentario